A vér mikrobiotájának és annak potenciális összefüggésének vizsgálata brojlereknél a bakteriális chondronecrosis és az osteomyelitis (BCO) között
Tárgyak
Absztrakt
Bevezetés
Anyagok és metódusok
Etikai nyilatkozat
Az ebben a vizsgálatban leírt összes állatmunkát az Arkansasi Egyetem Intézményi Állatgondozási és Felhasználási Bizottsága (IACUC) hagyta jóvá, és minden kísérletet a jóváhagyott irányelveknek és előírásoknak megfelelően hajtottak végre.
Állatkísérlet tervezés
Vérminta és DNS kivonás
Vérmintákat gyűjtöttünk 5 látszólag egészséges madárból tollban a 14., a 41. és a 49. napon a mikrobiota elemzéshez, 240 vérmintát kapva (5 madár tollon × 16 toll/mintavételi életkor × 3 mintavételi életkor). További 12 vérmintát gyűjtöttünk a BCO-nak engedő madarakról (n = 12; 5 és 7 minta 41, illetve 49 napos kortól), összesen 252 mintát elemezve 16S rRNS profilozás céljából. A vérmintákat aszeptikusan gyűjtöttük egy szárnyas vénából EDTA Vacutainers alkalmazásával. 1 ml vérmintát centrifugáltunk (5000 fordulat/perc 5 percig szobahőmérsékleten) mikrocentrifuga segítségével, és 200 μl buffy bevonatot gyűjtöttünk a steril lamináris áramlási kamrába, és -20 ° C-on tároltuk. A buffy rétegekből genomi DNS-t extraháltunk a BiOstic® Bacteremia DNS Isolation Kit (MoBio) segítségével a gyártó utasításai szerint. A DNS-mintákat Qubit 2.0 fluorométerrel (Life Technologies) elemeztük mennyiségük és tisztaságuk szempontjából, és -20 ° C-on tároltuk.
PCR protokoll a 16S rRNS gén amplifikációhoz
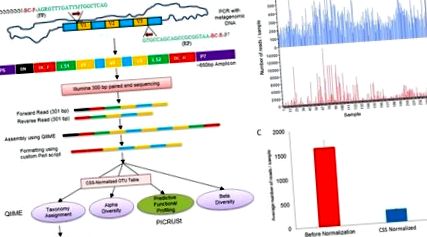
A 16S rRNS szekvenálásának sematikus stratégiája.
(a) A könyvtár előkészítését, a formázást a QIIME elemzés számára elfogadható leolvasási folyamatábrák mutatják be, amelyeket a LefSe és a PICRUSt alkalmazásával tovább elemeztek. * Normalizált OTU táblázatot használtunk a PICRUST elemzéshez. (FP = előreindító primer, RP = fordított primer, V = a 16s RNS gén variábilis régiója, P5 és P7 = Illumina szekvenáló primerek, N = véletlenszerű nukleotid, BC-F = előreindító vonalkódja, BC-R = fordított primer vonalkódja és LS = linker szekvencia). (b) Oszlopdiagram, amely 252 minta eloszlását mutatja be az OTU táblázat előtt (piros) és normalizálás után (kék). (c) Oszlopdiagram, amely a mintánként elolvasások átlagos számát mutatja standard hibával (piros) és a normalizálás után (kék).
Adatelemzés
A 16S rRNS-szekvencia elemzését felvázoló folyamatábra az 1a. Ábrán látható. Az Illumina szekvenálás előre és hátra párosított leolvasásait a Quantitative Insights into Microbial Ecology (QIIME 1.9.1 25) segítségével, a join_paired_ends.py szkript segítségével, fastq-join módszerrel állítottuk össze. Ezután az előreolvasott (BC-F) és a fordított leolvasási (BC-R) vonalkód-szekvenciákat összekapcsoltuk és összefogtuk (5'-vég) az összeállított szekvenciával (3'-vég), miután eldobtunk 8 nukleotid véletlenszerű szekvenciát és 27F és 533R primer szekvenciákat. egyedi Perl szkriptet használva, amely a QIIME használatával a downstream elemzéshez kompatibilis fastq fájlt készített.
Az olvasásokat az UCLUST 26 segítségével csoportosítottuk, és az operatív taxonómiai egységek (OTU) zárt referencia opcióval történő kiválasztását 13_8 GreenGenes adatbázis kiadással hajtottuk végre. Az OTU BIOM (biológiai megfigyelési mátrix) táblázatot QIIME-val (normalize_table.py) normalizáltuk kumulatív összeg-skálázási (CSS) módszerrel, amelyet azután taxonómiai hozzárendelésekhez, alfa-diverzitás becsléséhez, biomarkerek azonosításához és a metagenóm funkcionális tartalom előrejelzéséhez használtunk. Az alfa diverzitás indexeket egyirányú varianciaanalízissel (ANOVA) és post hoc az elemzést Tukey-Kramer HSD módszerrel végeztük. A hasonlóságok elemzését (ANOSIM) a metaadat-csoportok között súlyozatlan UniFrac távolságmérő segítségével végeztük el QIIME-val (Compare_categories.py). Ezenkívül a béta-változatosság becslésének fő koordinátanalíziseit (PCoA) QIIME-vel (24) végeztük, súlyozott és súlyozatlan UniFrac mérőszámok alkalmazásával, egyenletes mintavételi mélység mellett.
A biomarker azonosítását lineáris diszkrimináns elemzési hatásmérettel (LEfSe) 27 végeztük. A csirke vér mikrobiotájának metagenóm funkcionális tartalmát 16S rRNS génszekvenciák 28 felhasználásával PICRUSt-mal (Phenogenetic Investigation of Communities by Reconstruction of Unobserved States) jósoltuk. A baktériumhálózatot a Cytoscape 3.2.1 29 segítségével vizualizáltuk .
Eredmények
A QIIME demultiplexelés és a minőségi szűrés 4 153 965 összeállított szekvencia-leolvasást eredményezett 40 és 580 bp között, a medián hossza 469 bp, ami kimutató szekvenciák jelenlétét jelzi a rendellenes PCR-amplifikáció vagy a helytelen összeállítás miatt. Ebben a tanulmányban a zárt referencia OTU szedési módszert alkalmaztuk, mert egy új típusú mikrobiómát vizsgálunk, amelynek közösségi struktúrája, sokfélesége és tagsága nagyrészt ismeretlen. Ugyanakkor összehasonlítottuk a zárt referenciamódszer alkalmazásával kapott eredményeket mind a nyílt referencia, mind a de novo OTU szedési módszerekkel kapott eredményekkel (2. kiegészítő táblázat). Noha a nyílt referencia és a de novo OTU szedési módszer nagyobb számú OTU-t eredményezett (196. és 218. átlagnál), valamint mintánként magasabb olvasási értékeket eredményezett, az olvasások száma jelentősen nagy volt (átlagosan 72.5, illetve 79.3%). ), amelyeket a jelenlegi 13_8 GreenGenes adatbázis egyetlen rendszertani csoporthoz sem rendelt. Ezenkívül a zaj vagy kiméra szekvenciák hatékony eltávolítása érdekében a zárt referencia OTU-t választottuk a másik két módszer helyett.
A zárt referencia-OTU táblázat átlagos mintamélysége mintánként 1621 leolvasás volt (± 139,07), amint az az 1b. Ábrán látható. Az OTU táblázatot QIIME kumulatív összeg-skálázás (CSS) alkalmazásával normalizáltuk (1b. Ábra, c). Bár a minták átlagolvasása 6-szoros normalizálással csökkent (mintánként 266,55), ez a standard hibát is csökkentette (± 7,01). A CSS normalizált OTU táblázatot alkalmaztuk a taxonómia hozzárendeléséhez, az alfa változatosság becsléséhez, az LEfSe és a PICRUt elemzéshez. A béta változatosság elemzését különböző mintavételi mélységekben és ANOSIM-ban végeztük 999 permutációval, súlyozatlan UniFrac mérőszám alkalmazásával, mintánként 400 olvasás egyenletes mintavételi mélység mellett.
Rendszertani feladat
A taxonok eloszlása különböző szinteken.
Alfa sokféleség
Az alfa sokféleség a közösségen belüli sokszínűség mérése, és különböző mérőszámokat dolgoztak ki az alfa sokféleség mérésére, különös hangsúlyt fektetve a közösség szerkezetének különböző aspektusaira. Összességében a csirke vér mikrobiotája átlagosan 37,21 (± 1,13) OTU-t tartalmazott (min = 9; max = 107), 97% -os szekvencia-hasonlóság mellett (3. ábra; 3. kiegészítő táblázat). Jelentős kontrasztot tapasztaltunk a BCO és az egészséges csirke alfa változatosságában a PD teljes Tree index mellett (t-teszt, p 0,05). Ezenkívül az indító takarmányban lévő csirkék és a befejező takarmányok szignifikáns különbséget mutattak az alfa változatosságban (t-teszt, p 0,05) (3. ábra), ami összhangban van korábbi tanulmányunkkal 4 .
A CSS alfa-diverzitási indexe normalizálta a vér mikrobiota OTU táblázatát.
Béta változatosság
A hasonlóságok (ANOSIM) elemzése súlyozatlan UniFrac mutató alapján azt mutatta, hogy a BCO és az egészséges csirkék (R = 0,4402, p = 0,001) szignifikáns különbséget mutattak a baktériumközösség szerkezetében. A baktériumközösségek azonban nem különböztek szignifikánsan a csoportok között más szempontok szerint, ideértve a takarmány, a padló és a toll beállítását (1. táblázat). Az 1-gyel megegyező R-érték azt mutatja, hogy a minták teljesen különbözőek, míg a 0 azt jelenti, hogy azonosak. Fontos megjegyezni, hogy szignifikáns különbség mutatkozott az alfa változatosságban a BCO és az egészséges madarak között (PD egész fa index mellett) és a különböző korcsoportok között is (3. ábra). A súlyozott Unifrac távolságra épülő főkoordinátanalízis (PCoA) diagram megmutatja a BCO és az egészséges csirkék közötti különbségeket a közösségi struktúrában (4a. Ábra). A 41. nap baktériumközösségei szintén jellegzetesen különböztek a 14. és 49. napétól (4b. Ábra). Ez összhangban van korábbi megfigyeléseinkkel, miszerint a 41. nap közösségeinek alfa-diverzitási mutatói szignifikánsan magasabbak voltak a 14. és 49. naphoz képest. Ezenkívül a PCoA-sávban az első és a befejező takarmánnyal etetett csirkék között jól látható baktériumközösségek mutatkoztak (ábra (4c. Ábra), bár az ANOSIM nem tudta megragadni ezt a jelentős tulajdonságot (1. táblázat).
A csirkék vér mikrobiómáinak fő koordináta-analízis (PCoA) diagramja.
(a) Egészséges (n = 171) és BCO (n = 9) csirkék PCoA-diagramja súlyozott UniFrac mérőszám alkalmazásával, mintánként 400 leolvasási egyenletes mintavételi mélység mellett. (b) 14 napos (n = 30), 41 napos (n = 41) és 49 napos (n = 36) régi csirkék PCoA-diagramja súlyozott UniFrac mutatóval, mintánként 1000 leolvasási mélység mellett. (c) Az induló (n = 67) és a befejező (n = 154) étrendben táplált csirkék PCoA diagramja súlyozatlan UniFrac mérőszám alkalmazásával, mintánként 200 olvasásmélység mellett.
Az egészséges és a BCO csirkék baktériumközösségeiben tapasztalható jelentős különbséget hierarchikus csoportosítás is szemléltette. A 49 napos csirkék BCO csirkéit megkülönböztetve csoportosítottuk az egészséges csirkéktől. A 41 napos BCO csirkék azonban nem mutattak semmilyen jellegzetes klaszterképet (5a. Ábra). A csirkék (252 minta) és az OTU-k (baktériumfajok) közötti hálózati elemzés bizonyos fokú különbséget mutatott a BCO és az egészséges csirkék közötti interakciós mintákban (5b. Ábra), ami különböző baktériumközösségekre utal.
A csirkék csoportosulása a vér mikrobiotája alapján.
(a) Az egészséges és a BCO csirkék hierarchikus csoportosulása a vér mikrobiotáján alapul. A filogenetikai fát a FigTree V1.3.1 szoftver segítségével állítottuk elő, súlyozott UniFrac mérőszámmal, a normalizálás előtti adatok felhasználásával. A zöld és a piros vonalak a BCO csirkéket jelképezik 41, illetve 49 napos csirkékből. A fekete vonalak egészséges csirkék. A sor végén lévő szám a csirkeminta azonosítóját mutatja. (b) A Cytoscape V3.2.1 szoftverrel előállított csirkevér mikrobiota baktériumhálózata. A vörös, kék és fehér csomópontok a BCO csirkéket, az egészséges csirkéket és az OTU-kat jelentik. A zöld szegély a BCO csirkék hálózata, a narancs él pedig az egészséges csirkéké.
Észrevettük, hogy az összes BCO madár csak a két korcsoportból származott (41. és 49. nap) és két tollállományú csoport (W35–56 és W1–56). Ezért egy további béta-változatosság-elemzést végeztünk egy 77 minta részhalmazának felhasználásával, amelyek az adott korosztályhoz tartoznak, és a tollak felállított csoportjai, köztük 65 egészséges és 12 BCO madár. A súlyozott UniFrac metrikán alapuló ANOSIM elemzés nagyobb különbséget jelzett a BCO és az egészséges csirkék között (R = 0,5293, p = 0,001), mint a teljes adatsorral (n = 252). A 6. ábrán bemutatott PCoA-diagram szintén alátámasztja az ANOSIM-elemzés eredményét.
A csirkék kiválasztott részhalmazának vérmikrobiomáinak fő koordináta-analízis (PCoA) diagramja.
Egészséges (n = 51) és BCO (n = 9) csirkék PCoA-diagramja súlyozott UniFrac mérőszám alkalmazásával, mintánként 400 leolvasási egyenletes mintavételi mélység mellett.
A BCO biomarkerei
Az egészséges és a BCO csirkék között differenciálisan bőséges taxonómiai csoportokat lineáris diszkrimináns elemzési hatásmérettel (LEfSe) azonosítottuk α = 0,05, LDA pontszám legalább 2 és relatív bőség nagyobb, mint 0,1. Összesen 26 tulajdonság jelentősen eltérő volt az egészséges és a BCO csirkék között. Nemzetségi szinten a BCO csirkék vér mikrobiotáját differenciálva gazdagították nemzetségekkel Staphylococcus, Granulicatella és Microbacterium, mivel az egészséges csirkék gazdagodtak Pseudomonas, Enhydrobacter és Aquabacterium (7b. Ábra). Megfigyeltük azt is, hogy a menedékjog Firmicutes BCO csirkékben gazdagodott. Hasonlóképpen, Alphaproteobacteriumok nagyon gazdag volt BCO csirkékben, míg Betaproteobaktériumok és Gammaproteobaktériumok egészséges csirkéknél osztály szinten (7b. ábra).
LEfSe analízissel azonosított, differenciálisan bőséges taxonok, csirkék vér mikrobiotájával, relatív bőséggel ≥0,1.
(a) A taxonómiai cladogram LEfSe elemzés alapján készült. A vörös és a zöld BCO-val dúsított taxonokat, illetve egészséges csirkéket mutat. A fényerő arányos a taxonok bőségével. (b) A BCO csirkékben dúsított taxonok piros színnel jelennek meg, negatív LDA pontszámmal, egészséges csirkék zöld színnel, pozitív LDA pontszámmal (> 3,5 mindkét esetben). Két aláhúzás közötti taxon a GreenGene adatbázis javasolt neve.
A csirkevér mikrobiómák várható funkcionális genetikai kapacitása
A csirke vér mikrobiota előre jelzett funkcionális összetétele a PICRUSt alkalmazásával.
(a) A KEGG COG kategóriák relatív bősége az 1. szinten a csirkevér mikrobiómáiban. (b) A KEGG útvonal relatív bősége (≥0,5) a JMP szoftverrel létrehozott funkcionális útvonalak 3. szintű kategóriáiban. (c) Differenciálisan bőséges jellemzők (KEGG COG kategóriák, relatív bőség ≥0,5), amelyeket LEfSe alkalmazásával állítottak elő, LDA pontszám ≥2,8.
Vita
Ez az első átfogó tanulmány, amely nem emberi gerinces állatok vérében található baktérium mikrobiómákat elemez. Az utóbbi években egyre nagyobb az érdeklődés a test különböző szöveteihez kapcsolódó mikrobiota jellemzésére, különböző egészségügyi feltételek mellett. Az egykor sterilnek gondolt testrészeknek, mint például a vér 30, a gyomor 31, a hólyag 32, a tüdő 33, a csontok, az ízületek 4 és a 34 emlőnek megvan a bennük rejlő mikrobiota. A szöveti mikrobiota dysbiosis különböző betegségekhez kapcsolódik, beleértve a szív- és érrendszeri betegségeket 10, a cukorbetegséget 35, az alkoholmentes zsíros májbetegségeket (NASHD) 36, a gyulladásos bélbetegségeket (IBD) 37, a pikkelysömör 38-at, az elhízást 39, a gyermekkori asztmát 39, funkcionális bélbetegség 40 és vastagbélrák 41. Itt megvizsgáltuk a csirkék vér mikrobiotáját azzal a céllal, hogy azonosítsuk a BCO-val kapcsolatos potenciális bakteriális biomarkereket.
Az ebben a tanulmányban végzett adatelemzés eredménye azt mutatta, hogy a brojlercsirkék vérében 30–40 OTU-ból álló baktériumközösségek léteznek, életkortól és egyéb környezeti vagy gazdaállapotoktól függetlenül. A béta-változatosság elemzése (4a. És 6. ábra), hierarchikus klaszteranalízis (5a. Ábra) és baktériumhálózati elemzés (5b. Ábra), amelyek a vér mikrobiotáján és a hierarchikus klaszterezésen alapulnak, és a vér mikrobiota előre jelzett metagenómáján alapulnak (8b. Ábra). A BCO-madarak vérében található baktériumközösségek megkülönböztethetők az egészséges madarakétól, ami bizonyos szelektív nyomások jelenlétére utal, amelyek hozzájárulnak a BCO-madarak vérmikrobiómáinak eltolódásához. Azonban csak a PD teljes fája mutatott szignifikáns különbséget az alfa változatosságban a BCO és az egészséges madarak között, ami összhangban van a BCO madarak filogenetikailag távoli vér mikrobiotájával, amint azt a béta változatosság elemzés mutatja (4a. És 6. ábra).
Publikálatlan kísérőtanulmányunk során ugyanabból a nyájból származó vérmintákat közvetlenül gazdag agar táptalajra szélesztettük. Az eredmény azt mutatta, hogy a kolóniák száma a 49. napon folyamatosan magasabb volt a huzalpadlókon (L35W és W56), mint az alomban (L56), jelezve, hogy a huzal padlóján a stressz elősegítette a bakterémiát, valószínűleg a bél hámjának bakteriális transzlokációján keresztül. Ezenkívül a telepek száma a 49. napon szintén folyamatosan magasabb volt a béna madaraknál, mint az egészséges madaraknál. Ezek az eredmények erős korrelációt jeleznek a drót padlóburkolata által okozott stressz, a bakterémia súlyossága és a BCO sántaság között.
E tanulmány egyik gyakorlati alkalmazása olyan baktériumos biomarkerek azonosítása volt, amelyek felhasználhatók az egyes brojlercsirkék azonosítására a korábbi életkorban, amelyek hajlamosak a BCO fejlődésére a későbbi korokban. A tanulmány adatelemzése valóban különböző szintű taxonómiai csoportokat azonosított, amelyek jelentősen gazdagabbak a BCO madarakban, mint az egészséges madarak. Érdekes módon a nemzetség Staphylococcus egyike annak a 18 tulajdonságnak (köztük 3 nemzetség), amelyek jelentősen gazdagodtak a BCO mintákban, ami kiemeli ennek a nemzetségnek a fontosságát, amelyet gyakran izoláltak a 3,4,5 BCO elváltozásoktól. Nemrégiben Al-Rubaye et al. 44 számolt be, hogy a brojlerek kihívása S. agnetis, amelyet leggyakrabban izoláltak Staphyloccous A BCO légiókból származó fajok tanulmányukban szignifikánsan megnövelték a sántaságot 10-ről (kontroll) 40% -ra, míg egy másik izolátummal Enterococcus faecalis csökkentette a sántaság előfordulását. Jelölheti a S. agnetis mint a BCO patogenezis kórokozója a brojlercsirkéknél, bár a 16S rRNS génprofilozási adataink nem szolgáltattak érdemi információt a Staphylococcus fajok a taxonómiai kiosztás korlátozott felbontása miatt.
Ebben a tanulmányban azonban a BCO-t csak a 41 és 49 napos madaraknál mutatták ki, és ezért a baktériumok biomarkereinek korlátozott értéke van a BCO-ra hajlamos madarak korai diagnosztizálásában. Ezt a szempontot gondosan figyelembe kell venni a jövőbeni vizsgálatok kísérleti tervezése során, hogy lehetővé tegyék a potenciális baktériumos biomarkerek azonosítását fiatal madarak vérmintáiban, amelyek előrejelzik a BCO fejlődését idősebb korokban.
Az ebben a vizsgálatban elemzett vér mikrobiómák jelentős hatással vannak a brojlercsirkék egészségi állapotára, beleértve a BCO patogenezist, amint azt a tanulmány kimutatta, valamint a brojlercsirkék egyéb betegségeit vagy stresszállapotait.
további információ
Hogyan lehet idézni ezt a cikket: Mandal, R. K. et al. A vér mikrobiotájának és annak potenciális összefüggésének vizsgálata brojlereknél a bakteriális chondronecrosis és az osteomyelitis (BCO) között. Sci. ismétlés. 6., 25882; doi: 10.1038/srep25882 (2016).
- Az ABO vércsoport és a testtömeg-index szövetsége keresztmetszeti vizsgálat egy ghánai származású embertől
- Bakteriális endocarditis
- Bakteriális gastroenteritis MedlinePlus Medical Encyclopedia
- Bakteriális gasztroenteritis Johns Hopkins orvostudomány
- Baktériumterápia és mitokondriális terápia SpringerLink