Az extracelluláris mátrix gének variációja a testsúlycsökkenés utáni súlyvisszanyeréshez kapcsolódik nemspecifikus módon
Nadia J. T. Roumans
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás A Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Roel G. Vink
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás A Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Marij Gielen
Komplex Genetika Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Maurice P. Zeegers
Komplex Genetika Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Claus Holst
Egészségügyi és Társadalmi Központ, Prevenciós Orvostudományi Intézet, Koppenhágai Egyetemi Kórház, Hovedvejen 5, Frederiksberg, 2000 Koppenhága, Dánia
Ping Wang
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás A Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Biokémiai Genetikai Laboratórium, Klinikai Genetikai Tanszék, Maastrichti Egyetemi Orvosi Központ, Maastrichti Egyetemi Kórház, P. Debyelaan 25, 6229 HX Maastricht, Hollandia
Arne Astrup
Táplálkozás, Testmozgás és Sport Tanszék, Természettudományi Kar, Koppenhágai Egyetem, Nørre Allé 51, 2200 Koppenhága, Koppenhága, Dánia
Wim H. Saris
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás A Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Armand Valsesia
Nestlé Egészségtudományi Intézet, EPFL Innovációs Park, 1015 Lausanne, Svájc
Jörg Hager
Nestlé Egészségtudományi Intézet, EPFL Innovációs Park, 1015 Lausanne, Svájc
Marleen A. van Baak
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Edwin C. M. Mariman
Humánbiológiai Tanszék, NUTRIM Táplálkozási és Transzlációs Kutatás Metabolizmusban, Maastrichti Egyetem, Universiteitssingel 40, 6200 MD Maastricht, Hollandia
Társított adatok
Absztrakt
Elektronikus kiegészítő anyag
A cikk online verziója (doi: 10.1007/s12263-015-0506-y) kiegészítő anyagot tartalmaz, amely az engedélyezett felhasználók számára elérhető.
Bevezetés
Anyagok és metódusok
A résztvevők és a tanulmányterv
A nullával egyenlő vagy alacsonyabb pontszám azt jelezte, hogy a résztvevő fenntartotta vagy folytatta a fogyást (WM) a követési időszak alatt, míg a nullánál magasabb pontszám azt jelezte, hogy a résztvevő visszanyerte a súlyát (WR) a 6 hónapos követés során.
DNS kivonás és genotipizálás
A genotipizáláshoz a DNS extrahálásához EDTA-vér Buffy rétegeket használtunk. A genotipizálást az Illumina Bead Station System (IlluminaInc) segítségével végezte az IntegraGen az Illumina 660quad chip segítségével, amely mintánként 660 000 SNP-t és CNV-t elemez. Az összes SNP-re elvégeztük a QC genotípust, és az SNP-ket kizártuk az elemzésből, ha 2-es hívássebesség-értékük> 0,2 volt, hogy meghatározzuk, hogy a periosztin-SNP-k kapcsolódási egyensúlyhiányban vannak-e. A kötési egyensúlyhiány szerkezetét SNPStats alkalmazásával értékeltük (Sole és mtsai 2006).
A többváltozós lineáris regresszió visszafelé történő eliminációját alkalmazták annak ellenőrzésére, hogy az SNP-k kombinációi javíthatják-e az eredményt. Ehhez a férfi populációban megfigyelt négy jelentős SNP-t használták.
Genotípuselemzések: logisztikai regressziós elemzést alkalmaztunk az öröklés legjobb genetikai modelljének megtalálásához, amely leírja a jelentős SNP-k genotípusainak hatását. A modell akkor tekinthető a legalkalmasabbnak, ha a legalacsonyabb Akaike információs kritérium (AIC) pontszámmal rendelkezik, és ha ez az érték legalább 2-rel alacsonyabb, mint a többi modell. Ha több modell hasonlóan alacsony AIC-értékekkel rendelkezik, mint ezek a modellek ugyanolyan jól illeszkednek, akkor a logisztikus regressziót, az esélyhányadost (OR) és a 95% -os konfidencia intervallumot (95% CI) számítottuk ki, hogy meghatározzuk egy adott genotípus súlyvisszanyerés kockázatát. A genotípuselemzéseket Stata 12.0 (StataCorp LP) alkalmazásával végeztük.
Eredmények
A résztvevők jellemzői
Az értékek átlag ± SD. Az alanyokat négy csoportra osztjuk: a férfi testsúly-fenntartók (WM, n = 59), a súly-visszanyelők (WR, n = 100) és a női WM (n = 135) és WR (n = 175). A súlyfenntartási pontszámot (WMS) a 6 hónapos ad libitum diéta végén számítják ki: (súly 6 hónapos ad libitum diéta után - súly az LCD után)/(súly az alapvonalon - súly az LCD után)
WC derék kerülete, LCD alacsony kalóriatartalmú étrend, WMS súlymegtartó pontszám
* P # P 2, és az összes SNP eredményét az S1 táblázat mutatja. A kisebb allélfrekvencia (MAF) további kiválasztása csak 6 SNP-ben eredményezett 5% -nál magasabb allélfrekvenciát (a 2. táblázatban félkövérrel feltüntetve): rs7323378, rs9315503, rs9547947, rs2158836, rs12589592 és rs2672826. Az SNP-k közül három a periosztin génben és annak környékén található (POSTN, rs7323378, rs9315503, rs9547947), a többi SNP pedig a laminin-β1-ben (LAMB1, rs2158836), fibulin-5-ben (FBLN5, rs12589592) és kollagénben, XXIII, alfa1 (COL23A1, rs2672826) gének. A három POSTN SNP összekapcsolódási egyensúlyban volt: rs7323378 – rs9547947 (r 2 = 0,760), rs7323378 – rs9315503 (r 2 = 0,487) és rs9547947 – rs9315503 (r 2 = 0,370). Ez azt jelzi, hogy ezek a variánsok szorosan kapcsolódnak egymáshoz, ezért a további elemzéshez csak a legalacsonyabb P értékkel rendelkező SNP-t használták, amely a POSTN rs7323378. Női alanyok esetében 1 SNP maradt az SNP-k kiválasztása után, amelyeknek kisebb allélfrekvenciája meghaladja az 5% -ot: rs17516906. Ez az SNP a fibronektin 1 (FN1) génben található.
2. táblázat
Az egyes SNP allélokkal, mint prediktorokkal végzett regresszióanalízisek és a testsúly-fenntartási pontszámok, mint eredmények külön, a férfiak és a nők esetében
| Hímek | ||||||
| rs8031741 | EGY DOBOZ | 159 | 9.57E − 26 | 5% | ||
MAF kisebb allélfrekvencia
SNP többváltozós lineáris regressziós elemzések
A többváltozós lineáris regresszióban a férfipopulációban megfigyelt szignifikáns SNP-kkel történő visszafelé történő elimináció szignifikanciát eredményezett három SNP esetében: rs2672826 (β = −17.52, P = 0.020), rs2158836 (β = −11.50, P = 0.039) és rs7323378 (β = -13,67, P = 0,009). Ez azt jelzi, hogy a COL23A1, a POSTN és a LAMB1 együttesen additívan hat a súlyszabályozásra.
Genotípuselemzések
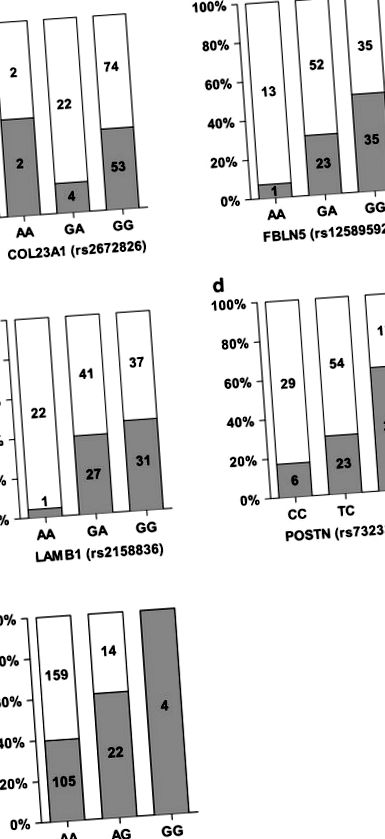
A szignifikáns SNP-k esetében az egyes genotípusok százalékos visszanyerési vagy fenntartási fenotípusa. Mindegyik oszlop a szignifikáns SNP-hez specifikus genotípussal rendelkező alanyok teljes mennyiségét mutatja: a COL23A1 (rs2672826), b FBLN5 (rs12589592), c LAMB1 (rs2158836), d POSTN (rs7323378), és e FN1 (rs17516906). hirdetés képviseli a férfiakat, és e képviseli a nőket. A szürke sávok a testsúly-fenntartók (WM) százalékát jelzik a genotípussal, a fehér sávok pedig a súly-visszanyerők (WR) értékét jelentik. Az egyes sávokon belüli szám az adott genotípussal rendelkező résztvevők száma
3. táblázat
Logisztikai regressziós elemzésekkel találták meg az öröklés legjobb genetikai modelljét, amely leírja a genotípusok hatását
| rs2672826 A> G COL23A1 | Együtt domináns | G/G G/A A/A | 1.00 3,94 (1,28–12,10) * 0,72 (0,10–5,25) | 206.4 |
| Uralkodó | G/G G/A – A/A | 1.00 2,86 (1,10–7,49) * | 206.6 | |
| Recesszív | G/G – G/A A/A | 1.00 0,59 (0,08–4,33) | 211.6 | |
| rs12589592 G> A FBLN5 | Együtt domináns | G/G G/A A/A | 1.00 2,26 (1,15–4,46) * 13.00 (1.61–104.81) * | 202.7 |
| Uralkodó | G/G G/A – A/A | 1.00 2,71 (1,40–5,25) * | 204.8 | |
| Recesszív | G/G – G/A A/A | 1.00 8.67 (1.10–68.06) * | 206.4 | |
| rs2158836 A> G LAMB1 | Együtt domináns | G/G G/A A/A | 1.00 1,27 (0,64–2,51) 18.43 (2.35–144.63) * | 199.3 |
| Uralkodó | G/G G/A – A/A | 1.00 1,89 (0,98–3,62) * | 210.1 | |
| Recesszív | G/G – G/A A/A | 1.00 16.36 (2.14–124.9) * | 197.8 | |
| rs7323378 T> C POSTN | Együtt domináns | T/T T/C C/C | 1.00 4,01 (1,85–8,67) 8,25 (2,85–23,88) * | 192.6 |
| Uralkodó | T/T T/C – C/C | 1.00 4,88 (2,35–10,16) * | 192.7 | |
| Recesszív | T/T – T/C C/C | 1.00 3,54 (1,37–9,14) * | 203.6 | |
| rs17516906 A> G FN1 | Együtt domináns | G/G A/G A/A | 1.00 0,00 (NA) 0,00 (NA) | 409,0 |
| Uralkodó | G/G A/G – A/A | 1.00 0,00 (NA) | 412.8 | |
| Recesszív | G/G – A/G A/A | 1.00 2,81 (1,40–5,63) * | 410.7 |
Az esélyhányadost, a 95% -os konfidenciaintervallumot, az Akaike információs kritériumot (AIC) és a P értékeket a logisztikus regressziós elemzésekből nyertük le a társdomináns, domináns és recesszív modellekkel. A legjobban illeszkedő modell rendelkezik a legalacsonyabb AIC-értékkel, és ez az érték legalább 2-rel alacsonyabb, mint a többi modell. P értékek (158K, pdf)
Köszönetnyilvánítás
Szeretnénk megköszönni a résztvevők együttműködését ebben a tanulmányban. Ezt a tanulmányt az NWO támogatta (TOP Grant 200500001).
Finanszírozás
Ezt a tanulmányt a Holland Szervezet a Tudományos Kutatásért finanszírozta, a támogatás száma: 200500001.
Az etikai normák betartása
Összeférhetetlenség
Nadia Roumans, Roel Vink, Marij Gielen, Maurice Zeegers, Claus Holst, Ping Wang, Arne Astrup, Wim H. Saris, Marleen A. van Baak és Edwin C.M. Mariman kijelenti, hogy nincsenek összeférhetetlenségük. Armand Valsesia és Jörg Hager teljes munkaidőben dolgozik a Nestlé SA-nál.
Etikai jóváhagyás
Az emberi résztvevők bevonásával végzett vizsgálatok során elvégzett összes eljárás összhangban volt az 1964-es Helsinki Nyilatkozattal és annak későbbi módosításaival vagy hasonló etikai normákkal. Ezt a tanulmányt az adott ország helyi etikai bizottságai hagyták jóvá: 1. a Maastrichti Egyetemi Kórház és a Maastrichti Egyetem, Hollandia Orvosi Etikai Bizottsága; 2. Dánia fővárosi régiójának orvosbiológiai kutatási etikai bizottságai; 3. Suffolk Helyi Kutatási Etikai Bizottság, Egyesült Királyság; 4. Krétai Egyetem Etikai Bizottsága, Görögország; 5. A Potsdami Egyetem Etikai Bizottsága; 6. Kutatási etikai bizottság a Navarrai Egyetemen, Spanyolország; 7. Az Endokrinológiai Intézet Etikai Bizottsága, Csehország; és 8. Etikai Bizottság a nemzeti közlekedési többprofilos kórházhoz Szófiában, Bulgáriában. Írásbeli tájékozott beleegyezést kaptak a vizsgálatba bevont minden egyes résztvevőtől.
- Az alma fogyasztásával járó fogyás Az American College of
- A súlykerékpározás magasabb halálozási kockázattal jár, tanulmány szerint a testsúlycsökkenés
- Fogyás Étkezési terv Mátrix, rutin a zsírvesztéshez - Dre Fitness
- A fogyás másik oldala Elena Lipson segítségével sajátítsa el stresszét, hogy visszanyerje egészségét
- A fogyás a kognitív funkció javulásával jár a túlsúlyos és elhízott emberek körében