Az RNS kölcsönhatásainak kémiai eltolódás feltérképezése a polipirimidin traktushoz kötődő fehérjével
Xuemei Yuan
Biológiai Tudományok Tanszék és Strukturális Biológiai Központ, Imperial Tudományos, Technológiai és Orvostudományi Főiskola, 1 Wolfson laboratórium, Exhibition Road, London SW7 2AY, Egyesült Királyság és 2 Blackett laboratórium, Prince Consort Road, London SW7 2BW, Egyesült Királyság
Natalia Davydova
Biológiai Tudományok Tanszék és Strukturális Biológiai Központ, Imperial Tudományos, Technológiai és Orvostudományi Főiskola, 1 Wolfson laboratórium, Exhibition Road, London SW7 2AY, Egyesült Királyság és 2 Blackett laboratórium, Prince Consort Road, London SW7 2BW, Egyesült Királyság
Stephen Curry
Biológiai Tudományok Tanszék és Strukturális Biológiai Központ, Imperial Tudományos, Technológiai és Orvostudományi Főiskola, 1 Wolfson laboratórium, Exhibition Road, London SW7 2AY, Egyesült Királyság és 2 Blackett laboratórium, Prince Consort Road, London SW7 2BW, Egyesült Királyság
Stephen Matthews
Biológiai Tudományok Tanszék és Strukturális Biológiai Központ, Imperial Tudományos, Technológiai és Orvostudományi Főiskola, 1 Wolfson laboratórium, Exhibition Road, London SW7 2AY, Egyesült Királyság és 2 Blackett laboratórium, Prince Consort Road, London SW7 2BW, Egyesült Királyság
Absztrakt
A polipirimidin traktus-kötő fehérje (PTB), egy homodimer, amely monomerenként négy RRM-típusú RNS-kötő domént tartalmaz, fontos szerepet játszik mind az alternatív splicing szabályozásában, mind a transzláció iniciálásának stimulálásában, bizonyos pikornavírusok belső riboszóma belépési helyei szerint . Kémiai eltolódási térképezési kísérleteket használtunk a PTB-34, a fehérje harmadik és negyedik RRM doménjét tartalmazó rekombináns fragmens és számos rövid pirimidinben gazdag RNS oligonukleotid közötti kölcsönhatások vizsgálatára. Az eredmények megerősítik, hogy az RNS-ek elsősorban a PTB-34 β-lapjának felületével lépnek kölcsönhatásba, ugyanakkor feltárják a fehérje-fragmensen belüli két hosszú rugalmas linker szerepét is, ezt az eredményt mutagenezis kísérletek is alátámasztják. A leképezés az RRM3 és az RRM4 különálló kötési preferenciáit jelzi, az előbbi különösen specifikus interakciót vált ki az UCUUC szekvenciával.
BEVEZETÉS
A polipirimidin-traktus-kötő fehérje (PTB) egy RNS-kötő fehérje, amelynek feladata a messenger és a pre-messenger RNS felhasználásának szabályozása számos különböző összefüggésben. Míg a PTB fő fiziológiai szerepe számos gén, például az α-aktinin, α- és β-tropomiozin, c-src, fibroblaszt növekedési faktor receptorok és a a GABAA receptor (1–11), a fehérje a poliadeniláció (12,13) és az mRNS lokalizációjának (14) szabályozásában is szerepet játszik. Ezenkívül a PTB-t számos pikornavírus veszi fel a transzláció megindulásának stimulátoraként, amelyet belső riboszóma belépési helyek (IRES) vezérelnek (15–20). A legújabb munka azt sugallja, hogy a fehérje szabályozhatja a hepatitis C vírus transzlációját azáltal, hogy kölcsönhatásba lép mind a vírusos RNS 5'-, mind 3'-végével (21,22), és stimulálhatja az APAF-1 sejtes IRES aktivitását. (23).
A PTB mindezekben a rendszerekben úgy működik, hogy kölcsönhatásba lép az RNS-szel és más kiegészítő fehérjékkel (splicing és transzláció iniciációs faktorok), bár a hatásmechanizmusok továbbra is homályosak. Az intronban lévő PTB-kötőhelyek és az IRES RNS megfigyelhető rövid pirimidin-motívumok ismétlődéseiben (pl. UCUU, UCUUC, UUCUCU, CUCUCU) (1,3,8,11,24), amelyek gyakran, de nem kizárólag, egy pirimidinben gazdag háttér. Ezeket a megfigyeléseket támasztják alá in vitro szelekció (8,25) és kötési kísérletek (26).
A PTB homodimer (26) és négy RRM-típusú RNS-kötő domént tartalmaz monomerenként (27). A deléciós mutagenezis vizsgálatok feltérképezték az elsődleges RNS-kötő aktivitást az egyes monomerek harmadik és negyedik RRM doménjére, bár az RRM1 és az RRM2 hozzájárulása szintén jelentős lehet (17,26,28–30). Úgy tűnik, hogy az RRM2 fontos szerepet játszik a PTB dimerizálásában (26,28). A PTB harmadik és negyedik RRM doménjét tartalmazó monomer fragmens, a PTB-34 oldatszerkezete feltárta, hogy az RRM3 atipikus szerkezettel rendelkezik az ilyen típusú modulokhoz, mivel az RNS-kötő funkcióként működő négyszálú β-lap felületét egy szál (29) meghosszabbította. A PTB-34 szerkezetét két kiterjesztett és rugalmas linker polipeptid is uralja, az egyik (17 aminosav) összeköti az RRM3 4. és 5. β-szálát, a másik pedig (25 aminosav) összeköti a két RRM domént, jelezve, hogy a fehérje nagyfokú konformációs variabilitása van, legalábbis az RNS-sel való kölcsönhatása előtt.
A PTB és RNS célpontok közötti kölcsönhatások szerkezeti vizsgálataink kiterjesztése érdekében kémiai eltolódási térképezési kísérleteket végeztünk a PTB-34 és számos rövid, szintetikus, pirimidinben gazdag RNS molekula közötti komplexeken. Ez a kísérleti megközelítés feltárja azokat az aminosavakat, amelyek érintkezésben vannak az RNS ligandummal. A különböző RNS-oligonukleotidokkal megfigyelt aminosav-érintkezések különböző eloszlásainak összehasonlítása új meglátásokat tár fel a PTB szekvencia-specifitásával kapcsolatban.
ANYAGOK ÉS METÓDUSOK
NMR spektroszkópia
15 N-jelölt PTB-34-et, amely az emberi PTB-1 335–531-es maradékát tartalmazza, Escherichia coli-ban expresszáltuk és a korábban leírt módon tisztítottuk (29). Szintetikus RNS-oligonukleotidokat készítettünk és géltisztítottunk a Dharmacon Research, Inc.-vel. Az NMR-térképezési kísérletekhez az RNS-oligonukleotidokat 0,5 mM 15 N-jelzett PTB-34-et tartalmazó oldatokban titráltuk 20 mM nátrium-acetátban, pH 5,4. 15 N-1 H HSQC spektrumot (31) RNS: PTB-34 mólarányban 0,5: 1, 1: 1 és 2: 1 arányban rögzítettünk, hogy megkönnyítsük a fehérjéhez való RNS-hez való kötődés által zavart rezonanciák követését és hozzárendelését. Az összes NMR-spektrumot 302 K hőmérsékleten, z-árnyékolt gradienssel és hármas rezonancia szondával felszerelt négycsatornás Bruker DRX500 alkalmazásával nyertük. Az NMR-adatokat NMRPipe/NMRDraw (32) alkalmazásával dolgoztuk fel, és NMRView (33) alkalmazásával elemeztük.
Mutagenezis és RNS-kötési kísérletek
A hisPTB-34a mutánsait (a humán PTB-1 324–531-es maradékai) átfedő PCR-rel állítottuk elő kidolgozott protokollok felhasználásával és expresszáltuk E. coliban (29). Az összes mutációt cDNS-szekvenálással igazoltuk. A PTB – RNS-kötési kísérleteket az EMCV IRES (17) 1. doménjének α-32 P-jelölt RNS-transzkriptumainak felhasználásával végeztük nitrocellulóz-szűrő kötési vizsgálatokban (29). Röviden: PTB – RNS-kötési reakciókat (75 µl) készítettünk és 24 ° C-on inkubáltunk legalább 15 percig 10 mM HEPES-ben (pH 7,25), 100 mM KCl-ban, 3 mM MgCl2-ban, 5% glicerinben, 1 mM DTT-ben, 50 µg/ml élesztő-tRNS (Boehringer Mannheim), 50 µg/ml humán szérumalbumin (Delta Biotechnology). Az RNS-koncentrációt tipikusan 4 nM-nál rögzítettük. A vizsgálatokat proteinkötő Protran BA-85 nitrocellulóz membrán (Schleicher és Schuell) felhasználásával végeztük. A membránt alaposan mossuk 10 mM HEPES-ben (pH 7,25), 3 mM MgCl2-ban, 5% glicerinben, 1 mM DTT-ben, és egy 96 lyukú dot-blotterre (Bio-Rad) rögzítjük. 65 µl kötési reakció felvitele előtt és után a membránt 180 µl mosópufferrel mostuk. A kísérletet követően a membránt megszárítottuk, és a megkötött PTB – RNS komplex mennyiségét Cerenkov-sugárzás szcintillációs számlálásával határoztuk meg. Eltérő rendelkezés hiányában az összes reagenst a Sigma-Aldrich cégtől szereztük be.
EREDMÉNYEK
Az RNS oligonukleotidok kiválasztása
A természetesen előforduló PTB-kötőhelyek rövid pirimidin-szekvenciák többszöri ismétléséből állnak, amelyek intron vagy IRES RNS szakaszába vannak ágyazva, amely általában> 100 nt hosszú. Az ilyen nagy célpontok nem alkalmasak az NMR elemzésére, ezért tanulmányaink a rövid motívumok megkötésére összpontosítottak. A kiválasztott szekvenciák hossza 4-10 nt között volt, és a korábban PTB-kötőhelyeken azonosított motívumokat vagy ezek kis variációit tartalmazta (táblázat (1. táblázat).
Asztal 1.
| 1 | UCUU a | Caspase-9 (4); klatrin könnyű lánc B (11); kalcitonin/kalcitáló génnel rokon peptid (13); fibroblaszt növekedési receptor-1 (51); GABA γ2 alegység (1); N-metil-d-aszpartát-receptor (11); a-aktinin (5); a-tropomiozin (8); β-tropomiozin (6) |
| 2 | CUUC | |
| 3 | UCUUC | |
| 4 | UCUUU a | Fibroblaszt növekedési receptor-2 (2) |
| 5. | UCUCU | Caspase-9 (4); klathrin könnyű lánc B (11); c-src (52); GABA γ2 alegység (1); a-tropomiozin (8); β-tropomiozin (6) |
| 6. | C UCUCU | c-src (52); α-tropomiozin (8) |
| 7 | UCCUCUUC | Mint fent az UCUUC esetében |
| 8. | UCUU CUCU | b Kaspáz-9 (4); klatrin könnyű lánc B (11); c-src (52); GABA γ2 alegység (1); a-tropomiozin (8); β-tropomiozin (6) |
| 9. | UCUU C UCUUC |
Félkövérrel kiemelve az UCUU szekvenciákat; Az UCUCU szekvenciák aláhúzva.
a A kémiai módosítástól és az RNáz T1 hasításától az EMCV és FMDV IRES szekvenciákban védett helyeken azonosított motívumok (24,44).
b Hivatkozások az UUCUCU hexamerikus motívum előfordulására vonatkoznak.
Az RNS-kötőhely feltérképezése a PTB-34-en
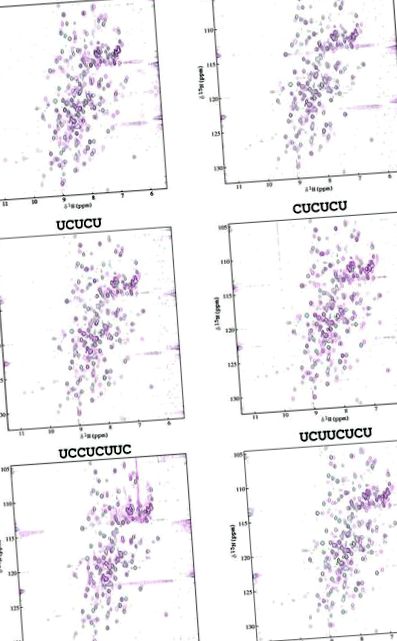
Az 1. ábra az 1. ábra mutatja az egyenletesen 15 N-jelzett PTB-34 HSQC spektrumainak átfedéseit, azonos oligonukleotidok esetén azonos mólmennyiségű ligandum RNS-sel és anélkül. Meg kell jegyezni, hogy az RRM3-ban a 4 és 5 β-szálakat összekötő, nagyon rugalmas hurokhoz tartozó hosszú rezonanciák vagy a hosszú, rugalmas tartományok közötti linker nem volt hozzárendelhető az apo-fehérjében a spektrum közepén tapasztalható súlyos átfedés miatt (29 ). Ezek közül sok kémiai eltolódási változást mutat az RNS-hez való kötődéskor, de a komplexek háromdimenziós NMR-adatait nem sikerült rögzíteni (a szekvencia-specifikus hozzárendelés megkönnyítése érdekében), mert az RNS-oligonukleotidok 8 órán belül előidézték a fehérjeminta lebomlását.
A 15 N-PTB-34 ± RNS 15 N-1 H HSQC spektrumának átfedése pH = 5,38, 302 K értéken rögzítve. Az apo-PTB-34 spektrumait fekete színnel, a holo-PTB-34 bíbor színnel mutatják.
A PTB-34 gerinc 15 N és 1 HN rezonanciájának kombinált kémiai eltolódási perturbációja (Δδ) RNS megkötésekor. A 15 N és 1 HN kémiai eltolódás abszolút értékeit ezen egyenlet segítségével kombináljuk: Δδ (15 N + 1 HN) = │Δδ 15 N│/4,69 + │Δδ 1 HN│. A 15 N kémiai eltolódás változásának korrekciós tényezőjét a 15 N spektrális szélességének és az 1 H spektrális szélességének arányaként kell kiszámítani (46,47). A 0,1 ppm a küszöböt vízszintes vonal jelzi.
A PTB-34 aminosavmaradékainak áttekintése, jelentős kémiai eltolódási változásokkal az RNS megkötésekor. A maradványok kombinált perturbációs eltolódása ≥0,1 ppm a fehérjéhez kötődő hat RNS oligonukleotid közül legalább négy zöld vagy narancssárga árnyalatú. A zöld árnyékolás azt jelzi, hogy az aminosav oldallánc oldószerrel van kitéve; az eltemetett oldalláncok narancssárga árnyalatúak. A hexamerikus RNP-2 és az oktamerikus RNP-1 motívumok mindkét RRM-ben dobozosak. A csillagok a legtöbb RRM szekvenciában aromás aromás csoportokat jelölik (48). A ’(3. ábra) jelöléssel ellátott maradványok mutációja. 3). A PTB figyelemre méltó az aromás maradékok hiányának hiányában, amely megtalálható a legtöbb más RRM fehérjében, és fontos egymásra épülő kölcsönhatásokat hoz létre az RNS bázisokkal. Azonban a hidrofób maradékok közül, amelyek a PTB-ben foglalják el ezeket az általában konzervált aromás pozíciókat (Leu 340 és Leu 378 az RRM3-ban, a Met 493 az RRM4-ben), jelentős kémiai eltolódásváltozás tapasztalható, jelezve, hogy fenntartják szerepüket az RNS-kötésben. Ezek az alifás oldalláncok a PTB-ben hasonló kölcsönhatásba léphetnek a nemi letalitás és a HuD kristályszerkezeteiben megfigyelt hidrofób Ile-bázis kontaktusokkal (35,36). Bár a PTB-ben maradt három „aromás” pozíció (Asn 376 az RRM3-ban, His 457 és Leu 495 az RRM4-ben) csak kismértékű vagy elhanyagolható kémiai eltolódási változásokat mutat RNS jelenlétében, ez nem zárja ki az RNS-kölcsönhatásokban való részvételüket. Valójában megfigyelték, hogy az His 457 mutációja csökkenti az RNS kötődését (29).
A különböző RNS oligonukleotidokkal összefüggő kémiai eltolódások mintázatának összehasonlítása számos érdekes különbséget tár fel. Az UCUUC szekvencia kémiai elmozdulásokat indukál, amelyek szinte kizárólag az RRM3 kötőfelületére csoportosulnak, jelezve, hogy ez az oligonukleotid specifikusan megköthető az RRM3-hoz (4. ábra. 4. ábra). Ezt az értelmezést alátámasztja az a megfigyelés, hogy az RNS dekamer (UCUUCUCUUC, oligonukleotid 9), amely az UCUUC szekvencia két másolatát tartalmazza, jelentős vonal szélesedést eredményez az egész spektrumban (az adatokat nem mutatjuk be), ami jelzi a magas molekulatömeg kialakulását. súlykomplexum, valószínűleg 1: 1-nél nagyobb RNS-sztöchiometriával. Ennek egyszerű magyarázata az lenne, hogy a PTB-34 két molekulája elsősorban az RRM3 moduljaikon keresztül kötődik az oligonukleotidhoz. Az UCUUCUCU oligonukleotid, amely csak annyiban különbözik a 9 oligonukleotidtól, hogy a 3 'végén nincs 2 nt, nem okoz azonos dimerizációt, megerősítve azt a következtetést, hogy az UCUUC két példányára van szükség egynél több PTB-34 molekula megkötéséhez. az oligonukleotid.
Az UCUUC szekvencia kisebb módosításai az UCUUU és az UCUCU előállításához drámai módon megváltoztatják a kémiai eltolódások eloszlását, nagyobb perturbációs felületet eredményezve, amely kiterjed az RRM4 β-lemezfelületének nagy részére (4. ábra (4. ábra). ). A poli (A) -val kötődő fehérje (PABP) N-terminális RRM doménpárjának kristályszerkezete a poli (A) -val komplexálva feltárta, hogy az RNS erősen kiterjesztett konfigurációban kötődik, így 6 nt elegendő az RRM pár átfedésére (37) Az UCUUU és az UCUCU pentamerek hasonló kiterjesztett konfigurációkat fogadhatnak el, amikor a PTB-34-hez kötődnek, ezt az értelmezést alátámasztja az a megfigyelés, hogy a kémiai eltolódás változásai 1: 1 RNS: PTB-34 mólaránynál közel voltak a maximálishoz. Mindazonáltal lehetséges, hogy ennek a két oligonukleotidnak az eloszlásának eloszlása a ligandumok áthelyezésének tudható be a két RRM doménen belül a β-lemez felületeken, így a megfigyelt perturbációs felület a különböző kötődési konformációk átlagát jelenti. Az RRM3-on és az RRM4-en átnyúló elmozdulások mintázatát a hexamer (CUCUCU) esetében is megfigyelték, amely nagyon hasonló perturbációs felületet mutat, mint az UCUCU (lásd az 1. és 4. ábrát). 4). Úgy tűnik, hogy az 5'-végén lévő extra pirimidin (citozin) nem befolyásolta a kötési specifitást.
A két oktamer, az UCCUCUUC és az UCUUCUCU mindegyike tartalmazza az UCUUC motívumot, de az 5'- és a 3'-végén további három pirimidinmaradék található. Az UCCUCUUC kémiai eltolódási zavarainak nagysága lényegesen kisebb, mint az UCUUC esetében (2. ábra, 2. ábra), ami azt jelenti, hogy az UCCUCUUC kisebb konformációs változásokat vált ki a fehérjében, vagy további kémiai eltolódást okoz több konformáció esetén. Tehát úgy tűnik, hogy az UCUUC szekvencia RRM3 kötési specifitása sérült az UCCUCUUC-ban. A másik oktamer UCUUCUCU esetében az RRM3 konformációs zavarának nagysága hasonló az UCUUC-hoz; de emellett az RRM4-ben az UCUCU pentameréhez hasonló nagyságrendű és specifitású változásokat is okoz (4. ábra, 4. ábra), amelyek megegyeznek az UCUUCUCU 3 'végén lévő öt maradékkal.
VITA
KÖSZÖNETNYILVÁNÍTÁS
Köszönetet mondunk Philip Sharpnak (MIT, USA), Richard Jacksonnak és Ann Kaminskinek (Cambridge-i Egyetem, Egyesült Királyság), Graham Belshamnek (IAH, Pirbright, Egyesült Királyság) és Doug Black-nek (UCLA, USA) a reagensekért és a megbeszélésekért. Ezt a munkát a Wellcome Trust és a biotechnológiai és biológiai tudományok kutatási tanácsának támogatása finanszírozza.
- Az RNS kölcsönhatásainak kémiai eltolódási térképezése a polipirimidin traktusba kötődő Nukleic fehérjével
- A FlaF egy β-szendvicsfehérje, amely rögzíti az Archaellumot az Archaeal sejt borítékába kötéssel
- Tudja meg, hogy a magas fehérjetartalmú étrend és a hajhullás összefüggenek-e a hajklubbal
- Arany fehérje energia harap
- Goldthread használat, mellékhatások, kölcsönhatások, adagolás és figyelmeztetés