Krónikus Hepatitis C kezelésben naiv betegek NS5A fehérjében a rezisztenciával összefüggő szubsztitúciók prevalenciáját befolyásoló tényezők
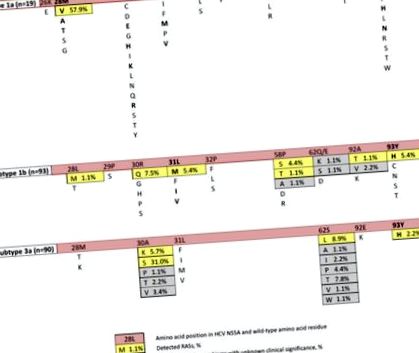
A hepatitis C vírus (HCV) NS5A aminosav polimorfizmusainak teljes listája, amely közvetlen hatású antivirális szerekkel szembeni rezisztenciához kapcsolódik (DAA). A félkövér vastagsággal társított szubsztitúciók (RAS) a virológiai kudarcok> 10% -ában vannak kiválasztva (a közzétett adatok alapján [3, 4, 5, 7, 9, 13, 18, 19].
Filogenetikai fa HCV 1a NS5A szekvenciákhoz (1126 nukleotid, 6330–7455. Pozíció a H77 HCV 1a referencia törzs szerint (GenBank NC004102)), GTR modell alapján felépítve; a 90% -nál nagyobb csoportmegbízhatóságú ágakat piros színnel jelölik; Az ebben a vizsgálatban szereplő HCV 1a szekvenciákat kék színnel jelöltük; az RAS (M28V) vagy a vad típusú (WT) típusát minden szekvenciához feltüntetjük. A japán HCV1a szekvenciákat [20] zöld színnel jelöltük. Az intravénás kábítószer-használóktól elkülönített szekvenciák, amelyek IDU-ként vannak jelölve.
RAS nélküli HCV 1b-hez épített NS5A-szekvenciák aminosav-kovariancia hálózatai (n = 72) (a); HCV 1b, legalább egy RAS-szal (n = 21) (b); HCV 3a RAS nélkül (n = 51) (c); HCV 3a legalább egy RAS-szal (n = 39) (d); HCV 1a (n = 19) (e); HCV 1a Japánból (n = 26) (f). A HCV-szekvenciák egymáshoz igazított aminosav-kovarianciáit ábrázoltuk a kovariációs pozíciókkal (csomópontok), amelyek körökként, és a pozíciók (élek) közötti kovarianciákkal vonalakként. A kovariancia hálózatok a ‘Fastcov’ nevű algoritmus segítségével épültek fel a golang (https://golang.org) programozási nyelvben (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4958985/). A futtatható bináris fájlok a következő címen érhetők el: http://yanlilab.github.io/fastcov. A hálózatokat az igraph R csomag segítségével tettük láthatóvá.
A HCV 1a, 1b és 3a vad típusú NS5A epitópiás profiljai, amelyek előrejelzik a CD8 + T sejt epitópok lokalizációját az orosz (a) és „globális” (c) szekvenciákban, valamint a CD4 + T sejt epitópok orosz (e) és „ globális ”(g) szekvenciák; A HCV 1a, 1b és 3a epitópiás pontszámainak különbsége RAS-mal és RAS nélkül RAS-hordozó régióban aa 20 és 100 között CD8 + T sejt epitópok esetében orosz (b) és „globális” (d) szekvenciák, valamint CD4 + T sejt epitópok között orosz (f) és „globális” (h) szekvenciákban a RAS pozícióit kiemelik a gyémánt görbék. Az előrejelzéseket a NetMHCpan-4.0 és a NetMHCIIpan-3.2 alkalmazásával végezték, és az Allele Frequency Net adatbázisból kivont globálisan elterjedt HLA-A, HLA-B, HLA-C és HLA-DR molekulák és 5 HLA-DP és 6 alapján készültek. HLA-DQ molekulák. Epitópprofilokat készítettünk minden HCV altípushoz úgy, hogy az egyes fehérjepozíciókhoz kiszámítottuk az adott HLA-t átfedő, előre jelzett HLA-kötő peptidek számát, az adott HLA gyakoriságával mérve és normalizálva úgy, hogy a maximális érték 1 legyen az elemzés minden egyes szakaszában.
Absztrakt
A hepatitis C vírus (HCV) NS5A aminosav polimorfizmusainak teljes listája, amely közvetlen hatású antivirális szerekkel szembeni rezisztenciához kapcsolódik (DAA). A félkövér vastagsággal társított szubsztitúciók (RAS) a virológiai kudarcok> 10% -ában vannak kiválasztva (a közzétett adatok alapján [3, 4, 5, 7, 9, 13, 18, 19].
Filogenetikai fa HCV 1a NS5A szekvenciákhoz (1126 nukleotid, 6330–7455. Pozíció a H77 HCV 1a referencia törzs szerint (GenBank NC004102)), GTR modell alapján felépítve; a 90% -nál nagyobb csoportmegbízhatóságú ágakat piros színnel jelölik; Az ebben a vizsgálatban szereplő HCV 1a szekvenciákat kék színnel jelöltük; az RAS (M28V) vagy a vad típusú (WT) típusát minden szekvenciához feltüntetjük. A Japánból származó HCV1a szekvenciákat [20] zöld színnel jelöltük. Az intravénás kábítószer-használóktól elkülönített szekvenciák, amelyek IDU-ként vannak jelölve.
RAS nélküli HCV 1b-hez épített NS5A-szekvenciák aminosav-kovariancia hálózatai (n = 72) (a); HCV 1b, legalább egy RAS-szal (n = 21) (b); HCV 3a RAS nélkül (n = 51) (c); HCV 3a legalább egy RAS-szal (n = 39) (d); HCV 1a (n = 19) (e); HCV 1a Japánból (n = 26) (f). A HCV-szekvenciák egymáshoz igazított aminosav-kovarianciáit ábrázoltuk a kovariációs pozíciókkal (csomópontok), amelyek körökként, és a pozíciók (élek) közötti kovarianciákkal vonalakként. A kovariancia hálózatok a ‘Fastcov’ nevű algoritmus segítségével épültek fel a golang (https://golang.org) programozási nyelvben (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4958985/). A futtatható bináris fájlok a következő címen érhetők el: http://yanlilab.github.io/fastcov. A hálózatokat az igraph R csomag segítségével tettük láthatóvá.
A HCV 1a, 1b és 3a vad típusú NS5A epitópiás profiljai, amelyek előrejelzik a CD8 + T sejt epitópok lokalizációját az orosz (a) és „globális” (c) szekvenciákban, valamint a CD4 + T sejt epitópok orosz nyelven (e) és „ globális ”(g) szekvenciák; A HCV 1a, 1b és 3a epitópiás pontszámainak különbsége RAS-mal és RAS nélkül RAS-hordozó régióban aa 20 és 100 között az orosz (b) és „globális” (d) szekvenciák CD8 + T sejt epitópjai, valamint a CD4 + T sejt epitópok között orosz (f) és „globális” (h) szekvenciákban a RAS pozícióit kiemelik a gyémánt görbék. Az előrejelzéseket a NetMHCpan-4.0 és a NetMHCIIpan-3.2 alkalmazásával végezték, és az Allele Frequency Net adatbázisból kivont globálisan elterjedt HLA-A, HLA-B, HLA-C és HLA-DR molekulák és 5 HLA-DP és 6 alapján készültek. HLA-DQ molekulák. Epitópprofilokat készítettünk minden HCV altípushoz úgy, hogy az egyes fehérjepozíciókhoz kiszámítottuk az adott HLA-t átfedő, előre jelzett HLA-kötő peptidek számát, az adott HLA gyakoriságával mérve és normalizálva úgy, hogy a maximális érték 1 legyen az elemzés minden egyes szakaszában.
- Biomedicines Free Full-Text Method Standardization for Naturalate Preference Studies
- Állatok ingyenes teljes szövegű anyai származás és hét orosz kecskepopuláció sokszínűsége
- Biomolekulák nélküli teljes szövegű étrendi nitrát a céklaléből a magas vérnyomásért Szisztematikus áttekintés
- Biomolekulák szabad teljes szövegű csökkent helyi válasz a kortikoszteroidokra az eozinofil krónikus betegségben
- Az állatok ingyenes teljes szövegű étkezési preferenciák a macskákban Az étrendi összetétel és az önmagukra gyakorolt hatása