A 2-metilhopanoid termeléséhez szükséges metiláz azonosítása és az üledékes hopánok értelmezésének következményei
Paula V. Welander
Biológiai Tanszék, Massachusettsi Műszaki Intézet, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
Maureen L. Coleman
Biológiai Tanszék, Massachusettsi Műszaki Intézet, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
Alex L. Sessions
b Földtani és Bolygótudományi Osztály, Kaliforniai Műszaki Intézet, Pasadena, MC100-23, 1200 East California Boulevard, Pasadena, CA 91125;
Roger E. Megidézi
c Föld, Légköri és Bolygótudományi Tanszék, Massachusetts Institute of Technology, 77 Massachusetts Avenue, E25-633, Cambridge, MA 02139; és
Dianne K. Newman
Biológiai Tanszék, Massachusettsi Műszaki Intézet, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
c Föld, Légköri és Bolygótudományi Tanszék, Massachusetts Institute of Technology, 77 Massachusetts Avenue, E25-633, Cambridge, MA 02139; és
d Howard Hughes Orvostudományi Intézet, Massachusetts Avenue 77, 68-171, Cambridge, MA 02139
Társított adatok
Absztrakt
Eredmények
A C-2 metilációhoz szükséges gén meghatározásához először feltételezett hopanoid bioszintetikus géneket azonosítottunk az R. palustris TIE-1 genomjában. Jelenleg az egyetlen véglegesen ismert hopanoid bioszintetikus lépés a szkvalén kezdeti ciklizálása az alapvető hopénszerkezet diplopténjává a szkvalén-hopén cikláz (shc) által. A Zymomonas mobilis és a Bradyrhizobium japonicum korábbi vizsgálata több feltételezett hopanoid bioszintézis gént azonosított az shc lokusz körül (24). Megvizsgáltuk a TIE-1-ben az shc lókusztól felfelé és lefelé mutató régiókat, és találtunk egy hasonló feltételezett hopanoid bioszintetikus géncsoportot (1. A. ábra). Körülbelül kiló kilobázis (kb) a szkvalén-hopén-cikláz-géntől felfelé nézve nyitott leolvasási keretet (ORF 4269) figyeltünk meg, amelyet feltételezett B-12-kötő gyök SAM-fehérjeként jegyeztünk fel (1. ábra A). Mivel kimutatták, hogy az R. palustris sejtek a C-2 helyzetben jelölt 2-metilhopanoidokat termelnek jelzett metioninnal táplálva (20, 25), ésszerűnek tűnt, hogy ez a metiláció S-adenozil-metionin (SAM) függő mechanizmus révén történhet. Egy radikális SAM motívum jelenléte az ORF 4269-ben tehát vonzó jelöltet tett a metilázra.
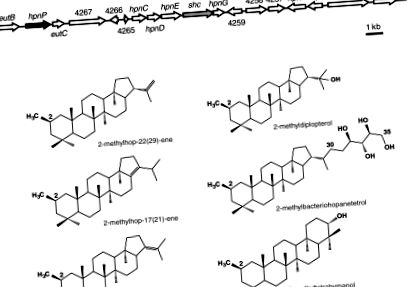
Egy feltételezett hopanoid bioszintetikus géncsoport azonosítása az R. palustris TIE-1-ben. (A) A hopanoid bioszintézishez szükséges shc lokuszt (Gray Arrow) több feltételezett hopanoid bioszintézis gén veszi körül. Az shc előtt egy hipotetikus B-12 kötő gyök SAM fehérjét (hpnP, Black Arrow) azonosítottak C-2 metiláz jelöltként. (B) Az R. palustris TIE-1 által előállított hat C-2 metilezett triterpenoid szerkezete: három 2-metilhopén (hop-22 (29) -én, hop-17 (21) -én és hop-21- ene), 2-metildiplopterol, 2-metiltetrahymanol és 2-metilbakteriohopanetetrol.
Az ORF 4269-et homológ rekombináció által közvetített géndelécióval töröltük a keretből, és az összes lipid-kivonatot gázkromatográfia-tömegspektrometriával (GC-MS) vizsgáltuk metilezett hopanoidok előállítására. Kimutatták, hogy a R. palustris TIE-1 legalább hat különböző C-2 metilezett triterpenoidot termel (1. B ábra), és az így létrejött deléciós törzs nem hozta létre ezeket a molekulákat (2. ábra és S1. Ábra). A C-2 metilezett hopanoidok termelését helyreállítottuk úgy, hogy a R. palustris metiláz gén egy példányát a natív promoter alatt önreplikáló plazmidon láttuk el (2. ábra). Ezért az ORF 4269 az egyetlen olyan gén, amely a metilációhoz szükséges a C-2 helyzetben, és javasoljuk, hogy a lokuszt átnevezzék hpnP-re, a Z. mobilis és a B. japonicumban megállapított nomenklatúra alapján (24).
R. palustris tenyészetek acetilezett összes lipidkivonatainak GC-MS teljes ionkromatogramjai. A ΔhpnP mutáns nem termel semmilyen C-2 metilezett hopanoidot. (A) R. palustris TIE-1. (B) R. palustris ΔhpnP. (C) R. palustris ΔhpnP a hpnP génnel kiegészítve egy önreplikálódó plazmidon. Számozott vegyületek: I, 2-metilhopének; II, hopének; III, 2-metildiplopterol; IV, diplopterol; V, 2-metil-tetrahymanol; VI, tetrahymanol; VII, 2-metilbakteriohopanetetrol; és VIII., bakteriohopanetetrol. Az acetilezett összes lipidkivonatokat magas hőmérsékletű GC-MS-sel elemeztük, és a vegyületeket a retenciós idők és a tömegspektrumok hiteles vegyületekkel való összehasonlításával és a közzétett tömegspektrumokkal azonosítottuk (S6. Táblázat). A II csúcs két hopénszerkezet, a hop-22 (29) -én és a hop-21-én együttelúciója. Nem metilezett hopanoidok eluálódnak 29–42 perc között (teljes kromatogram az S1. Ábrán látható). A komplementált AhpnP törzs 2-metilbakteriohopanetetrolt (VII vegyület) termelt, amint azt a 205 Da tömegkromatogram és a tömegspektrum elemzésével igazoltuk; a csúcs azonban túl széles ahhoz, hogy a GC-MS teljes ionáramának ezen ábrája felbontásánál látható legyen.
Szkvalén-hopén cikláz (shc) és hpnP metiláz gének előfordulása távoli rokonságban lévő baktériumokban. A fa filogenetikai kapcsolatokat képvisel a baktériumfajok között, és maximális valószínűséggel konstruálták öt összefűzött magfehérje (Adk, RpoB, GyrB, RecA és SecY) felhasználásával. A belső gyűrűben található kék sávok shc törzseket jeleznek; A külső gyűrűben található vörös sávok a hpnP törzseket jelzik. Az összes shc-t és/vagy hpnP-t tartalmazó szekvenált izolátumot, valamint más baktériumcsaládok képviselőit mutatjuk be. A Bacillus (34 izolátum) és a Burkholderia (58 izolátum) nemzetségek összeomlottak.
Amint az az S1 táblázatból kiderül, a metilázt tartalmazó organizmusok sokféle környezetben találhatók, beleértve az édesvizet, a talajt, a szennyvizet, a szárcsomókat és a gyökércsomókat. A tenyésztett tengeri cianobaktériumok 2-metilhopanoidjainak ritkaságával összhangban (17) a metiláz gént tartalmazó szekvenált cianobaktériumok közül egyiket sem izolálták tengeri környezetből. Mivel az üledékes 2-metilhopánok többségét vélhetően a fototrófok által uralt, sekély tengeri környezetből származnak (18, 19), arra számítottunk, hogy bioinformatikai elemzésünk feltárhatja a HpnP metiláz jelenlétét egy még nem tesztelt tengeri cianobaktériumban. Az α-proteobacterium Nitrobacter sp. Az Nb-311A volt az egyetlen tengeri organizmus mind a genomi, mind a tengeri metagenomikus adatbázisokban, amely tartalmazta a HpnP metiláz homológját.
A fehérje eredetének megértése érdekében rekonstruáltuk annak filogenitását a legnagyobb valószínűséggel, és teszteltük a topológia érzékenységét a bemeneti összehangolásra és a csoporton kívüli taxonok megválasztására. Az összes kísérlethez a ProtTest (30) által kiválasztott LG szubsztitúciós modellt (29) alkalmazták, és 10 véletlenszerű kiindulási fát használtak annak megakadályozására, hogy a fák keresése helyi optimumba szoruljon. Mivel az igazítás minősége meghatározó tényezője a fa pontosságának (31, 32), három igazítási programot használtunk (CLUSTALW, Muscle és T-Coffee) (33 –35). Ezenkívül három szűrési stratégiát (nyugodt, szigorú és szűrés nélküli) használtunk a bizonytalan beállítási pozíciók eltávolítására (S2. Ábra). Noha a szűrés növelheti az adatok jel-zaj arányát, elveti a potenciálisan informatív oldalakat is, és ez nem lehet előnyös rövid igazításoknál (36, 37). A fa gyökerezéséhez a radikális SAM fehérjék testvércsoportjának összes tagját alkalmaztuk outgroupként (S3. Ábra), amelyet optimálisnak javasoltunk (38). Összehasonlításképpen néhány kísérlethez hozzáadtunk egy második outgroup kládot (S3. Ábra).
A gyökér nélküli ingroup topológia a Methylobacterium fajokból, a Bradyrhizobium/Nitrobacter/Rhodopseudomonas csoportból és a cianobaktériumokból származó HpnP fehérje különálló csoportjait mutatja (S4. Ábra). Ezeknek a csoportoknak az elágazási sorrendje azonban jelentősen változott a vizsgálatok között. Három különböző gyökeres topológia jelent meg (4. ábra és S3. Táblázat). Leggyakrabban az α-proteobakteriális HpnP szekvenciák monofiletikusak voltak és a cianobaktériumok HpnP szekvenciáinak soraiba ágyazódtak be, és ebből a topológiából a leghevesebb arra következtetni, hogy az ős HpnP cianobaktériumban tartózkodott (4. ábra A). Bizonyos esetekben a cianobaktériumok és az α-proteobaktériumok szekvenciák testvértesteket képeztek (4. ábra B), vagy a cianobaktérium szekvenciák beágyazódtak az α-proteobaktérium szekvenciákba (4. ábra C). Ez a kétértelműség arra utal, hogy nincs elegendő filogenetikai jel ahhoz, hogy magabiztosan oldhassuk meg a főbb kládok elágazási sorrendjét. A megközelítőleg elfogulatlan teszttel teszteltük, hogy az adott összehangolás esetén helyreállított legjobb topológia lényegesen jobb-e, mint a két versengő topológia (39). Ez a teszt nem vetette el a versengő hipotézisek egyikét sem (S4. Táblázat), és így még egy adott igazítás esetén sem tudjuk magabiztosan kiválasztani a legjobb fát.
Három távoli rokonságban lévő baktériumfül HpnP-metilázainak filogenetikai összefüggései. (A) A 15. kísérletben talált legjobb fa (S3. Táblázat), korán elágazó cianobaktérium HpnP-vel. (B) A legjobb fa a kilencedik kísérletben, monofiletikus cianobaktérium HpnP-vel. (C) A második fa legjobb fája, korán elágazó Methylobacterium HpnP-vel. Az elágazási támogatást nem paraméteres közelítő valószínűségi arányteszt (aLRT) statisztikák jelzik. A nemzetség rövidítései: M = Methylobacterium, N = Nitrobacter, B = Bradyrhizobium és R = Rhodopseudomonas.
A pontos elágazási sorrendtől függetlenül eredményeink a HpnP komplex evolúciós történetét tárják fel. Filetikai eloszlása kétféleképpen merülhet fel: vagy a cianobaktériumok és az α-proteobaktériumok őseiben volt jelen, és ismételten elveszett, vagy vízszintesen került a cianobaktériumok és az α-proteobaktériumok közé. Géntranszfert figyeltek meg ezen phyla között (40), és a cianobaktériumok leggyakrabban osztják meg a géneket az α-proteobaktériumokkal, összehasonlítva a többi phylával (41). A cianobaktériumokon belül a HpnP szórványosan oszlik el, és a HpnP filogenitás nem egyezik meg a filogenitás fajokkal (42). Ez magában foglalja a géntranszfert és a génveszteséget a cianobaktériumok között, így lehetetlen általánosítani, hogy az összes ősi cianobaktérium termelt-e 2-metilhopanoidokat. A magányos acidobaktérium helyzete a HpnP fában szintén bizonytalan. Az α-proteobaktériumokon belül a HpnP és a fajok filogenikái hasonlóak (43), ami függőleges süllyedést jelent ezen a csoporton belül. Ebből a filogenetikai elemzésből kiderül, hogy a HpnP fa kitöltéséhez szükséges több taxon megtalálása kritikus fontosságú e fehérje történetének jobb feloldásához.
Vita
Ebben a tanulmányban azonosítottuk a hpnP által kódolt gyökös SAM-metilázt, amely a bakteriális hopanoidok C-2-metilezéséhez szükséges. Jelenleg úgy tűnik, hogy a hpnP gén robusztus előrejelzője a 2-metilhopanoidok termelő képességének; hogy továbbra is exkluzív prediktor marad-e, az még várat magára, mivel a jövőben több metiláz gént azonosítanak. Egyelőre azonban a 2-metilhopanoidok és az üledékes 2-metilhopánok használata a cianobaktériumok indikátoraként arra késztetett bennünket, hogy értékeljük a hpnP géntermék eloszlását a baktériumok genomjában. Megállapítottuk, hogy a HpnP jelenléte nem korlátozódik a cianobaktériumokra, mivel az α-proteobaktériumokban (különösen a rhizobiales) és egy acidobacteriumban is megtalálható.
Összefoglalva, négy bizonyíték azt sugallja, hogy a 2-metilhopánok nem használhatók biomarkerként az oxigénes fotoszintézishez: i) a hpnP sok modern baktériumon oszlik el, amelyek nem vesznek részt oxigénes fotoszintézisben, ii) a cianobaktériumok nem kötelező oxigénes fotoautotrófok (51), iii) nem minden cianobaktérium termel 2-metilhopanoidokat (17), és iv) a cianobaktérium Nostoc punctiforme 2-metilhopanoidjai az akinéták külső membránjain lokalizálódnak, amely túlélési struktúra nem fotoszintetikusan aktív (23). Ezért szükséges a 2-metilhopanoidok biológiai funkciójának feloldása a modern baktériumokban, hogy jobban megértsük a 2-metilhopánok jelentését az ősi üledékekben. Függetlenül sejtfunkcióiktól, a (phylo) genetika alkalmazása egy adott geostabil vegyület bioszintéziséhez szükséges gén történetének azonosítására és értelmezésére megmutatja, hogy a molekuláris kövületek két osztálya - a gének és a biomarkerek - miként korlátozhatják egymást és a mikrobák evolúciójának és anyagcseréjének megalapozottabb megértése.
Anyagok és metódusok
Baktériumtörzsek és növekedési körülmények.
Az ebben a vizsgálatban használt baktériumtörzseket az S5. Táblázat tartalmazza. Az Escherichia coli törzseket lizogén táptalajban (LB) növesztettük 37 ° C-on. A Rhodopseudomonas palustris törzseket kemoheterotróf módon növesztett YP táptalajban (0,3% élesztő kivonat, 0,3% pepton) növesztettük 30 ° C hőmérsékleten sötétben, miközben 250 RPM sebességgel rázattuk. Szilárd táptalajon történő növekedéshez az LB-t vagy az YP-t 1,5% agarral megszilárdítottuk, és szükség esetén 20 μg/ml (E. coli) vagy 800 μg/ml (R. palustris) gentamicinnel egészítettük ki.
DNS-módszerek, plazmidépítés, transzformáció és törzsépítés.
Az összes plazmidszerkezetet az S5. Táblázat ismerteti. QIAprep Spin Miniprep Kit-et (Qiagen) alkalmaztunk a plazmid DNS izolálásához E. coli-ból. Az R. palustris törzsek genomi DNS-ét a DNeasy Blood and Tissue Kit (Qiagen) alkalmazásával izoláltuk. Valamennyi klónozó köztitermék DNS-szekvenciáját szekvenciával igazoltuk az MIT Rákkutató Központ Biopolimer Laboratóriumában. Az E. coli törzseket elektroporációval transzformáltuk az Electroporator 2510 (Eppendorf) alkalmazásával, a szállító ajánlása szerint. A plazmidokat az E. coli S17-1-ből R. palustris-ba mozgattuk YP-agar lemezeken történő konjugációval, amelyeket egy éjszakán át 30 ° C-on inkubáltunk (52, 53). A hpnP lokusz törlését a TIE-1-ben és a deléciós mutáns kiegészítését a korábban leírtak szerint végeztük (15).
Gázkromatográfia-tömegspektrometriás (GC-MS) elemzés.
A hopanoid elemzéshez a törzseket késői álló fázisig növeltük (7 d). Az összes lipidkivonatot oldószeres extrakcióval állítottuk elő, acetátokká derivatizáltuk, és magas hőmérsékletű GC-MS-sel elemeztük, a korábban leírtaknak megfelelő DB-XLB oszlop alkalmazásával (15). A vegyületeket a retenciós idők és a tömegspektrumok összehasonlításával azonosítottuk autentikus vegyületekkel (tetrahymanol a Trimyema sp-től; diplopterol a Methylococcus capsulatus-tól; bakteriohopanepentol a M. capsulatus-tól) és publikált tömegspektrumokat [hopenes: (54); tetrahymanol: (55)]. A C-2 metilálást az m/z 191 és 205, m/z 369 és 383 közötti fragmens eltolódásából és a desmetil-homológhoz viszonyított relatív retenciós időből (~ 0,15 perccel korábban) következtettük. A retenciós időket és a molekuláris ionokat az S6. Táblázat tartalmazza.
A HpnP filogenitása.
A HpnP homológjait a BLAST segítségével azonosítottuk és ClustalW, Muscle vagy T-Coffee (33–35) alkalmazásával igazítottuk össze. Az igazításokat Gblocks (56) vágta szigorú (alapértelmezett paraméterek) vagy laza beállításokkal. A szigorú eset esetében egyetlen hézag sem volt megengedett sorrendben. A nyugodt esetben a konzervált és a szomszédos pozíciók szekvenciáinak minimális számát a minimálisan megengedett értékre, a nem konzerválatlan pozíciók maximális számát 100-ra, a blokk minimális hosszát 4-re, a szekvenciák felében pedig hézagokat engedték meg. Az S2 ábra az Muscle által generált és a Gblocks által levágott példát mutatja be. A két outgroup család megfelelőségét megerősítette a rokon fehérjék kimerítőbb fája (S3. Ábra), amely egyértelműen azt mutatja, hogy a HpnP és az outgroup családok szorosan kapcsolódnak egymáshoz, de eltérő kládok.
A maximális valószínűségű fákat a PhyML (29) állította össze [a ProtTest (30) által kiválasztott LG + gamma modell, 6 gamma sebességi kategória, 10 véletlenszerűen induló fa, az SPR + NNI elágazás cseréje és az adatok alapján becsült szubsztitúciós paraméterek felhasználásával. Az ágazati támogatást SH-szerű hozzávetőleges LRT statisztikák határozták meg (29). A fajfát hasonlóan készítették el öt összefűzött fehérjeszekvencia (RpoB, RecA, Adk, GyrB és SecY) felhasználásával 375 fajból, Gblocks alkalmazásával 1679 pozícióig vágva és Firmicutes-ben gyökerezve. A fajfát nem szándékozzák teljes körűnek tekinteni, hanem arra használják, hogy bemutassák az shc és hpnP gének megoszlását a baktériumok phylájában.
A fa topológiáinak összehasonlításához a CONSEL-ben (57) végrehajtott, megközelítőleg elfogulatlan tesztet (39) használtuk. A 4. ábrán bemutatott topológiákat használták korlátozásként, és ezekre a topológiákra PhyML optimalizálta az elágazás hosszát és a szubsztitúciós paramétereket. Ezután a legjobb kötetlen fa valószínűségét összehasonlítottuk a két alternatív fa valószínűségével, megadva az AU teszt p-értékét az egyes fákhoz (S4. Táblázat). Bármely fa, amelynek p-értéke kisebb, mint 0,05, elutasítható. Ha több fa p-értéke meghaladja ezt a határértéket, akkor nem tudjuk magabiztosan kiválasztani közülük az igaz fát.
Kiegészítő anyag
Köszönetnyilvánítás.
Köszönetet mondunk David Doughty-nak, Jacob Waldbauernek és Eric Almnak a technikai segítségért és a hasznos beszélgetésekért, valamint a bírálóknak a kéziratot javító konstruktív észrevételeikért. Ezt a munkát a Nemzeti Repülési és Űrkutatási Hivatal (NASA) Exobiológiai Programjának (ALS, DKN és RES), a NASA Asztrobiológiai Intézet (RES), a National Science Foundation Postdoctoral Minority Fellowship (PVW) és az Agouron Institute támogatásával támogatták. Geobiológiai Posztdoktori Ösztöndíj (MLC). D.K.N. a Howard Hughes Orvosi Intézet nyomozója.
Lábjegyzetek
A szerzők nem jelentenek összeférhetetlenséget.
- A simaizomsejtekből származó 130 kDa sejtfelszíni LDL-kötő fehérje azonosítása részben
- Hogyan konfigurálhatom a konzult termelési környezetben az Ubuntu DigitalOcean készüléken
- IJMS szabad teljes szövegű Aedes albopictus autofágia-kapcsolódó gén 8 (AaAtg8) szükséges a konferenciához
- Metabolikus hőtermelés - áttekintés a ScienceDirect témákról
- A nikotinsav nem megfelelő bevitele súlyos egészségügyi következményekkel járhat