Ketogén étrend és mikroRNS-ek kapcsolódnak az antioxidáns biokémiai homeosztázishoz
Roberto Cannataro
1 GalaScreen Laboratories, Mater Domini Kórház 88100 Catanzaro és Gyógyszerészeti, Egészségügyi és Táplálkozástudományi Tanszék, Calabria Egyetem, 87036 Rende (CS), Olaszország
Maria Cristina Caroleo
2 Gyógyszerészeti, egészségügyi és táplálkozástudományi tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Alessia Fazio
2 Gyógyszerészeti, Egészségügyi és Táplálkozástudományi Tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Chiara La Torre
2 Gyógyszerészeti, Egészségügyi és Táplálkozástudományi Tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Pierluigi Plastina
2 Gyógyszerészeti, egészségügyi és táplálkozástudományi tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Luca Gallelli
3 Klinikai farmakológiai és farmakovigilanciai operatív egység, Egészségtudományi Tanszék, Magna Graecia Egyetem, Mater Domini Kórház Catanzaro, 88100 Catanzaro, Olaszország
Graziantonio Lauria
2 Gyógyszerészeti, Egészségügyi és Táplálkozástudományi Tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Erika Cione
2 Gyógyszerészeti, Egészségügyi és Táplálkozástudományi Tanszék, Calabria Egyetem, Via Pietro Bucci, 87036 Rende (CS), Olaszország
Absztrakt
Nemrégiben bemutattuk a ketogén étrend (KD) képességét a mikroRNS (miR) expressziós profil befolyásolására. Itt arról számolunk be, hogy a KD képes normalizálni a miR expressziót elhízott alanyokban, összehasonlítva a sovány alanyokkal. Két különböző bioinformatikai eszköz alkalmazásával azt tapasztaltuk, hogy a sovány alanyokban összehasonlítható szintre visszatérő miR-ek közül négyen specifikusan kapcsolódnak az antioxidáns biokémiai utakhoz, a többiek pedig mind antioxidáns, mind gyulladáscsökkentő biokémiai útvonalakhoz. Különösen érdekes a hsa-miR-30a-5p újraszabályozása, amely korrelál a vörösvértestekben a kataláz expressziós fehérje csökkenésével.
1. Bemutatkozás
2. Anyagok és módszerek
2.1. Népesség
Az összes humán vizsgálat etikai jóváhagyását a Regionális Etikai Bizottság (REC) (# 120-18052018) szerint adták meg. Az alanyokat akkor is kizártuk, ha magas vérnyomás jelenlétét mutatták ki és/vagy gyógyszeres kezelésben részesültek. Úgy ítélték meg, hogy a tanulmány nem indított klinikai vizsgálatokat, és nem regisztrálták ilyenként. A résztvevőktől írásbeli, tájékozott beleegyezést kaptak, amely megfelelt az etikai gyakorlat normáinak, amelyeket a Helsinki nyilatkozat vázolt fel. A kizárási kritériumok közé tartoztak a cukorbetegség, a vesebetegségek, a májműködési zavarok, az alkohol- vagy drogfogyasztás története és a daganatos betegségek a tanulmányt megelőző öt évben.
2.2. Immunblot elemzés és RNS kivonás
Vérmintákat gyűjtöttünk, és az antitest (abcam # ab16731) segítségével gyűjtöttük a vörösvérsejteket és a kataláz (CAT) expressziós fehérje lizátumát. A minták fehérjekoncentrációját a [17,18,19,20] részben leírtak szerint becsültük meg. Ezután a szérum plazmát alkalmazták a funkcionális genomikai vizsgálatokhoz. A teljes RNS-t 200 μl vérszérumból vagy plazmából extraháltuk miRNeasy Serum/Plasma Kit (Qiagen, Venlo, Hollandia) segítségével a potenciális szennyező anyagok csökkentése érdekében [15,21] és a gyártó utasításainak megfelelően.
2.3. NanoString minta előkészítése és adatelemzés
A NanoString Technology n-counter flexjéhez 100 ng RNS/miR-t használtunk bemenetként. A miR-eket ezután egy nCounter Human-V3 miRNS expressziós assay CodeSet-rel hibridizáltuk egy éjszakán át 65 ° C-on és a [22, 23] -ben leírtak szerint. A megbízható eredmények elérése érdekében a szórás és az átlag arányát kifejező és százalékban kifejezett variációs koefficienst (CV) 30% -nak választottuk. A vér hemolízissel összefüggésben lévő miR-eket kizártuk az elemzésből [24,25].
2.4. In Silico A hsa-miR célgének előrejelzése
Annak érdekében, hogy a tömb elemzéséből a géneket a hsa-miR-ek célpontjaként azonosítsuk, in silico elemzést végeztünk. A célgének in silico azonosítását a miRTargetLink Human (https://ccb-web.cs.uni-saarland.de/mirtargetlink/) és a DIANA Tools (http://diana.imis.athena-innovation.gr) segítségével végeztük el. /DianaTools/index.php) adatbázisok. Ez utóbbi adatbázist használták annak ellenőrzésére, hogy mely miRNS célgének már kísérletileg validáltak.
2.5. Statisztikai analízis
Asztal 1
| Kor, y | 46,5 ± 10,51 | 46,83 ± 12,32 | 46,6 ± 11,56 | ns | ||||
| Magasság, cm | 175,1 ± 5,2 | 171,3 ± 6,6 | 176,3 ± 3,3 | ns | ||||
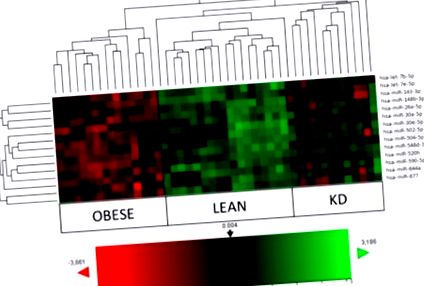
| Súly, kg | 107,5 ± 3,0 | 70,8 ± 3,8 | 96,97 ± 11,2 | 2 | 33,9 ± 1,2 | 22,1 ± 2,5 | 31,5 ± 1,3 | Az 1. ábra, beleértve a hsa-let-7b-5p, a hsa-miR-143-3p, a hsa-miR-148b-3p, a hsa-miR-26a-5p, a hsa-miR-502-5p, a hsa-miR aláírásait -520h, hsa-miR-548d-3p, hsa-miR-590-5p és hsa-miR-644a. Különösen KD az elhízott alanyokhoz viszonyítva normalizálta a hsa-let-7b-5 (8/12), a hsa-miR-143-3p (9/12), a hsa-miR-148b-3p (10/12), hsa-miR-590-5p (10/12), hsa-miR-520h (8/12) és hsa-miR-644a (9/12), amelyek az alanyok több mint 65% -ában fejeződtek ki, míg a hsa-miR-548d-3p esetében 100% -ot értek el (12/12). A hsa-miR-26a-5p (6/12) és a hsa-miR-502-5p (7/12) esetében legalább az 50% -ot elérték. A hsa-miR-504-5p esetében nem volt változás. Az itt azonosított új hsa-miR-let7e-5p (5/12) és hsa-miR-877 (5/12) 50% alatti jelenlétet mutatott, a hsa-miR-30a-5p (8/12) kivételével ). |
Az elhízott (n = 14), a sovány (n = 17) és a ketogén étrend (KD) alanyainak (n = 12) hőtérképe és hierarchikus csoportosítása a legkülönbözőbb módon expresszált mikroRNS-ek alapján. A dobozok színe és intenzitása a génexpresszió változását jelzi. Az elemzés során a piros a lefelé szabályozott géneket, a zöld pedig a felfelé szabályozott géneket jelenti. A fekete változatlan kifejezést képvisel, amint azt a színreferencia is mutatja. n-Solver szoftvert használtak.
3.3. A Silico-találatokban
Két különböző adatbázist használtak a szilikon elemzéshez. Az adatokat összehasonlítottuk a mindkét adatbázisban kísérletileg validált célgének számával. Az eredményeket a 2. táblázat tartalmazza. Bár hasonló eredményeket találtunk két hsa-miR esetében (félkövérrel a 2. táblázatban), a többi hsa-miR esetében talált validált célok száma következetesen különbözött. A DIANA eszközökben az validált célgének száma magasabb volt a miRtagertLink Human vonatkozásában; ezért DIANA eszközöket alkalmaztak a további bioinformatikai elemzésekhez.
2. táblázat
Bioinformatikai eszközök az in silico elemzéséhez.
| miRTargetLink Human | DIANA Tools | |
| hsa-let-7b-5p | 124 | 312 |
| hsa-let-7e-5p | 15 | 273 |
| hsa-miR-143-3p | 32 | 82 |
| hsa-miR-148b-3p | 10. | 218 |
| hsa-miR-26a-5p | 52 | 391 |
| hsa-miR-30a-5p | 119 | 458 |
| hsa-miR-30e-5p | 7 | 412 |
| hsa-miR-502-5p | 3 | 30 |
| hsa-miR-504-5p | 6. | 7 |
| hsa-miR-520h | 5. | 5. |
| hsa-miR-548d-3p | 1 | 203 |
| hsa-miR-590-5p | 2 | 43 |
| hsa-miR-644a | 2 | 0 |
| hsa-miR-877 | 0 | 19. |
3.4. Validált hsa-miR kölcsönhatás és a kataláz Western blot elemzése
A megjósolt és validált célgéneket a DIANA Tools segítségével értékeltük. Megállapítást nyert, hogy az új hsa-miR-let7e-5p szabályozza a glutation-peroxidázt 7 (GPX7), amint azt a húrelemzés (2. A ábra), valamint az in silico 3'UTR kölcsönhatás mutatja (2. B ábra). A tet metil-citozin-dioxigenáz 3 (TET3) in silico 3′UTR régiói a hsa-miR-520h (2. ábra D) interakcióhoz és a szuperoxid-diszmutáz 2 (SOD2) 3'UTR régiói a hsa-miR-548d-3p-hoz (2. ábra F ), valamint a húrelemzés a 2. ábrán C, E, ill. A húrelemzés azt mutatta, hogy 10 fehérje képes fizikailag kölcsönhatásba lépni a GPX7-gyel (2. B ábra), valamint a TET3-mal (2. D ábra), az SOD2-vel (2. F ábra) és a katalázzal (CAT) (3. D ábra). Ez utóbbi fehérjét Western blot analízissel követtük nyomon, amint az a 3. ábrán látható. Densitometrikus elemzést hajtottunk végre, és a CAT fehérje szintjének jelentős csökkenését mutattuk, amint azt a 3. B. ábra mutatja. Megállapítást nyert, hogy a hsa-miR-30a-5p A CAT 3'UTR régiói, amint azt a 3. C. ábra mutatja. Ugyanakkor azt találtuk, hogy más célgéneket is befolyásol a KD, és kapcsolódnak az antioxidáns anyagcseréhez és a gyulladással kapcsolatos génekhez, amint azt a 3. táblázat mutatja. A rövidítéseket és a génneveket a 4. táblázat ismerteti .
Bioinformatikai elemzés. A (A) GPX7 és (B) GPX7 in silico kölcsönhatások a 3′UTR régióval. A (C) A TET3 és in silico kölcsönhatásai a 3′UTR régióval (D). A (E) SOD2 és in silico kölcsönhatásai a (3′UTR) régióval (F).
- Az alacsony zsírtartalmú étrend a férfiak alacsonyabb tesztoszteronszintjéhez kapcsolódik EurekAlert! Science News
- Alacsony zsírtartalmú étrend alacsonyabb tesztoszteronszinttel összefüggésben Urology Times
- Nagy hatékonyságú Eco Spray fogyás vélemények Szisztematikus áttekintés Ketogén étrend fogyás Conquip
- 30 napig próbáltam a ketogén étrendet
- A ketogén étrend mellékhatásainak azonosítása Epilepszia Alapítvány