A tuberkulózis metagenomikus elemzése múmiában
A szerkesztőhöz:
1994-ben 242 holttestet tartalmazó kriptát fedeztek fel a magyarországi Vácon. Sok holttest természetesen mumifikálódott, beleértve Hausmann Terézia maradványait (a Kiegészítő Függelékben 68-as testként emlegetik, amely a levél teljes szövegével elérhető a NEJM.org oldalon), aki 1797. december 25-én, 28 évesen hunyt el. életkorától. 1,2 A maradványok mellkasi röntgenfelvétele tiszta volt, de a test cachektikus megjelenése összhangban volt a tuberkulózis diagnózisával. Egy korábbi tanulmányban a testből kapott mellkasi minta molekuláris elemzése megerősítette a tuberkulózis diagnózisát, és néhány korlátozott genotípusos és mennyiségi információt szolgáltatott, amelyek a mikobaktériumok DNS-jének rendkívül jó megőrzését javasolták. 1,2
A legtöbb korábbi kísérlet a mikobaktérium DNS visszanyerésére történeti vagy ősi mintákból a polimeráz-lánc-reakció (PCR) amplifikációjára támaszkodott, amely a termék hordozásának és szennyeződésének kockázatával társult. Alternatív módszer a hibridizációs befogás, 3 de a PCR-hez hasonlóan az ismert szekvenciák célzásán és az egyes törzsek vagy fajok vizsgálatainak nehéz fejlesztésén alapul.
A „metagenomika” kifejezés a tenyésztetlen mintákból kinyert DNS nyílt végű szekvenálására utal, célspecifikus amplifikáció vagy dúsítás nélkül. Ezt a megközelítést alkalmaztuk a DNS szekvenciák kimutatására Mycobacterium tuberculosis a múmiából nyert mintában.
A korábban leírt módszerekkel 1,2 kivontuk a DNS-t egyetlen tüdőszövetmintából, és a DNS-t egyetlen Illumina MiSeq futtatással szekvenáltuk a gyártó utasításainak megfelelően (módszereink és eredményeink teljesebb leírását lásd a Kiegészítő Függelékben. ).
5,5 millió páros végű olvasmányt kaptunk (a Sequence Read Archive-ban raktuk le SRP018736 csatlakozási számmal). A leolvasások kevesebb mint 1% -a igazodik az emberi genomhoz, míg 8% -uk a M. tuberculosis referencia H37Rv törzs; ez mintánként átlagos lefedettségi mélység 32 × volt M. tuberculosis genom (lásd a Kiegészítő Függeléket). Mert nem kulturáltunk M. tuberculosis vagy bármelyiket elvégezte M. tuberculosis–Specifikus PCR elemzések laboratóriumunkban, és mivel mély, sőt genomi lefedettséget értünk el, úgy véljük, hogy nagyon alacsony az esély arra, hogy artefaktív szekvenciákat kapjunk a szennyezés miatt.
1.ábra. 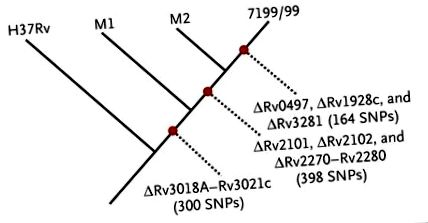
Az M1 és M2 törzsek elvesztették az Rv3018A – Rv3021c lókuszt, ezt a tulajdonságot a 7199/99 törzs is osztja. A H37Rv-hez képest mindhárom törzs 300 egy nukleotid polimorfizmust (SNP) oszt meg egymással. Az M1 és M2 között törlődik az Rv2101, Rv2102 és Rv2270 – Rv2280 lókusz. Az M2 és a 7199/99 (amely szorosan kapcsolódik a Haarlem és Erdman törzsekhez) további 398 SNP-t oszt meg, amelyek nincsenek jelen az M1-ben. A 7199/99 törzs további három deléciót és további 164 SNP-t tartalmaz, összehasonlítva az M2-vel.
Amikor összehasonlítottuk a metagenomikat M. tuberculosis a H37Rv-vel olvassa, bizonyítékot találtunk arra, hogy az illető kettővel vegyesen fertőzött M. tuberculosis genotípusok (lásd a Kiegészítő Függeléket). Korábbi spoligotipizálási vizsgálatok a Hausmann családból származó mintákban két genotípust azonosítottak, 1 és a korabeli tuberkulózis nemrégiben készült áttekintése rávilágított a vegyes törzsű fertőzések fontosságára, különösen azokon a területeken, ahol a tuberkulózis nagyon endémiás. 4 A törlési mintákból a M. tuberculosis a múmia metagenómája a H37Rv-hez képest arra a következtetésre jutunk, hogy a múmia mindkét törzse egy Németországban 1998 és 2010 között bekövetkezett járvány törzséhez hasonlít (S1 és S2 ábra a Kiegészítő Függelékben). 5 A múmia egyik törzse, az M2 jobban hasonlított a járványtörzshez (7199/99), mint a másik törzs, az M1 (1. ábra).
Ez a tanulmány megmutatja a metagenomikus elemzés erejét, ha információt szolgáltat történelmi és talán kortárs példányokból.
Jacqueline Z.-M. Chan, Ph.D.
Martin J. őrmester, Ph.D.
Warwicki Egyetem, Coventry, Egyesült Királyság
Oona Y.-C. Lee, Ph.D.
David E. Minnikin, D.Phil.
Gurdyal S. Besra, Ph.D.
Birminghami Egyetem, Birmingham, Egyesült Királyság
Pap Ilidkó, Ph.D.
Magyar Természettudományi Múzeum, Budapest, Magyarország
Mark Spigelman, Ph.D.
Tel-Avivi Egyetem, Tel-Aviv, Izrael
Helen D. Donoghue, Ph.D.
University College London, London, Egyesült Királyság
Mark J. Pallen, MD, Ph.D.
Warwicki Egyetem, Coventry, Egyesült Királyság
m. [e-mail védett] ac. uk
A szerzők által biztosított közzétételi űrlapok e levél teljes szövegével elérhetők a NEJM.org oldalon.
1. Fletcher HA, Donoghue HD, Taylor GM, van der Zanden AGM, Spigelman M. A Mycobacterium tuberculosis DNS molekuláris elemzése egy 18. századi magyar családból. Mikrobiológia 2003; 149: 143 - 151
2. Fletcher HA, Donoghue HD, Holton J, Pap I, Spigelman M. Mycobacterium tuberculosis DNS elterjedt előfordulása a 18.-19. Századi magyaroktól. Am J Phys Anthropol 2003; 120: 144 - 152
3. Bouwman AS, Kennedy SL, Muller R és munkatársai. A Mycobacterium tuberculosis történelmi törzsének genotípusa. Proc Natl Acad Sci U S A 2012; 109: 18511 - 18516
4. Cohen T, van Helden PD, Wilson D és mtsai. Vegyes törzsű Mycobacterium tuberculosis fertőzések és a tuberkulózis kezelésének és ellenőrzésének következményei. Clin Microbiol Rev 2012; 25: 708 - 719
5. Roetzer A, Diel R, Kohl TA és mtsai. Teljes genomszekvenálás a hagyományos genotipizálással szemben a Mycobacterium tuberculosis járvány kivizsgálására: longitudinális molekuláris epidemiológiai vizsgálat. PLoS Med 2013; 10: e1001387 - e1001387
- A lumbosacralis gerinc tuberkulózisának eredményei és kezelése 53 beteg retrospektív vizsgálata
- Az anya hüvelyi mikrobiómájához kapcsolódó koraszülés A hüvelyi baktériumok elemzése a
- Meta-elemzés a műszakos munkavégzésről és az egyes elhízási típusok kockázatairól - V - 2018 - Elhízásvélemények -
- Helyes ételválasztás a tuberkulózis esetén, amely állapot károsította Amitabh Bachchan máját
- A szülők véleménye a gyermekkori elhízásról a beszélgetőtábla hozzászólásainak kvalitatív elemzése Kortárs