A csecsemőknél a bél mikrobiota és a gazdaszervezet étrendfüggő interakciójának metagenomikai vizsgálata az immunválasz különbségeit tárja fel
Absztrakt
Háttér
A bél mikrobiota és a gazdaszervezet kölcsönös kapcsolatban állnak, a mikrobiota funkcionális összetétele erősen befolyásolja a gazdaszervezet egészségét és jólétét. Ezért fontos egy szintetikus megközelítés kidolgozása a gazda transzkriptóm és a mikrobiom egyidejű tanulmányozására. A csecsemőknél a korai mikrobiális kolonizáció kritikus fontosságú az újszülöttek bél- és immunfejlődésének irányítása szempontjából, és különösen vonzó az ember-kommensális kölcsönhatások kialakulásának tanulmányozása szempontjából. Itt beszámolunk a három hónapos, kizárólag anyatejjel és tápszerrel táplált csecsemők bélmikrobiómájának és a gazda hám transzkriptumának egyidejű vizsgálatának eredményeiről.
Eredmények
Változást figyeltek meg mind a gazda mRNS expressziójában, mind a mikrobiom filogenetikai és funkcionális profiljában a szoptatott és tápszerrel táplált csecsemők között. A gazda hámsejt-génexpressziója és a baktériumok metagenomikus alapú profiljai közötti kölcsönös összefüggés vizsgálatához a gazdaszervezet transzkriptóm és funkcionálisan profilozott mikrobiom adatait új, többváltozós statisztikai elemzéseknek vetettük alá. A bél mikrobiota metagenoma virulencia jellemzői egyidejűleg változtak az immunitáshoz kapcsolódó gén expresszióval a hámsejtekben a tápszerrel táplált és a szoptatott csecsemők között.
Következtetések
Adataink betekintést nyújtanak a gazda transzkriptóm és mikrobiom integrált válaszaiba az étrendi szubsztrátokra a korai újszülöttkori időszakban. Bebizonyítottuk, hogy az étrendbeli különbségek a bél kolonizációján keresztül befolyásolhatják a veleszületett immunrendszerhez kapcsolódó gének gazda expresszióját. Ezenkívül az ebben a tanulmányban bemutatott módszertan adaptálható más gazda-kommensális és gazda-patogén kölcsönhatások értékelésére genomi és transzkriptikus adatok felhasználásával, szintetikus genomikai alapú képet nyújtva a gazda-kommenzális kapcsolatokról.
Háttér
A bél mikrobiota mély hatással van a gazda egészségére és wellnessére. Például csíramentes malacokon végzett vizsgálatok egyértelműen szemléltetik a megváltozott bélnövekedést [1], az emésztési enzimaktivitást [2] és a bélhez kapcsolódó nyirokszövet fejlődését [3]. A metagenomikus, metatranscripticus és metaproteomikus analitikai technikák által lehetővé tett molekuláris szintű vizsgálatok átformálják annak megértését, hogy a bél mikrobioma miként modulálja a gyomor-bélrendszer morfológiai, immunfejlődését [1–4], a génexpressziót [5] és általában a gazda biológiáját. [6, 7]. Bár számos tanulmány kimutatta a diéta hatását a csecsemő mikrobiotájára [8–10], a fejlődő csecsemő bél és a kolonizáló mikrobiota genomjáról és transzkriptóm szintű keresztbeszéléséről keveset lehet tudni. Születéskor az emberi csecsemő bélrendszere funkcionálisan éretlen és steril. Ennek megfelelően a korai újszülött periódus kritikus fázis mind a bél emésztési fejlődésében, mind a kommensális mikrobiota által végzett kolonizációban.
Eredmények
A vizsgálatba összesen hat BF csecsemő és hat FF csecsemő édesanyát vettek fel. Röviden, minden csecsemőből székletmintákat gyűjtöttünk, és mikrobiális DNS-t extraháltunk és szekvenáltunk. Ezenkívül mRNS-t izoláltak a székletből, amely bélbél exfoliált hámsejteket tartalmazott, és feldolgoztuk a mikroarray elemzéshez [12]. Ez a két egyidejű művelet szolgáltatta a nyers mikrobiális metagenomikus és gazdaszervi transzkriptiás adatokat. Ezt követően egymástól függetlenül, majd egyidejűleg elemeztük a szekvencia és a mikroarray adatokat, majd azonosítottuk a bélhám transzkriptóm és a mikrobiális metagenom közötti többváltozós összefüggéseket. Az eljárást az 1. kiegészítő fájl ismerteti (a részletekért lásd: Anyagok és módszerek). Amint az 1. táblázat mutatja, a csecsemők és az anyák adatai megfelelő egyensúlyban voltak az FF és a BF csecsemők között.
A diéta hatása a gazda transzkripciós válaszaira
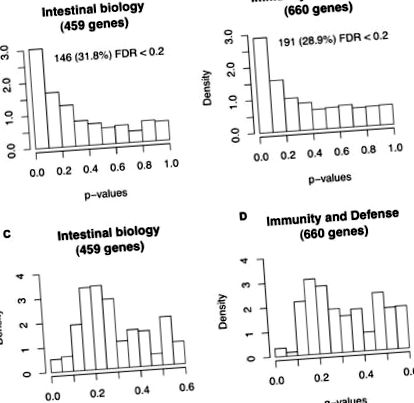
Amint azt a 2. kiegészítő fájl mutatja, általában az FF gazdasejt minták alacsonyabb génexpressziós értékeket mutatnak a BF gazdasejt mintákhoz képest. Ezek az adatok összhangban állnak egy rhesus majom-vizsgálattal, amelyben a tápszeres táplálás lefelé szabályozta a teljes bélgén expressziót a szoptatott majmokhoz képest [14]. Két adatállomány - 459 bélbiológiával kapcsolatos gén, valamint 660 immunitás és védekezéssel kapcsolatos gének - génjeit teszteltük a BF és FF csecsemők közötti differenciál expresszióra, hamis felfedezési arányú (FDR) [15] többszörös tesztkorrekciójú permutációs teszt alkalmazásával. Amint az az 1. ábrán látható, a várható gének eleve hogy reagálni tudjunk az étrendre, a differenciál expresszió érdekében dúsítottuk. Ez arra utal, hogy mi eleve a releváns gének kimutatását lehetővé tevő ismeretek. Utóvizsgálatként 459 bélbiológiai génből 146, 660 immunitás- és védekező génből 191 mutat FDR q-értéket 1. ábra
Az étrend hatása a bél mikrobiomjára
Rendszertani elemzés
Amint a 2a, b ábrán látható, lényeges különbségek voltak az azonosítható 16S rRNS taxonómiai eloszlásában az FF és a BF csecsemő mikrobiotájában. Az FF csecsemő mikrobiota nagyon homogén volt a menekültügyi szintű eloszlásokban. Pontosabban, körülbelül azonos arányban voltak Firmicutes és Actinobacteriumok (egyenként körülbelül 40%), a fennmaradó baktériumok túlnyomórészt proteobaktériumokból álltak. Egy FF csecsemő mikrobiota egyértelműen kiugrott, és az Actinobacteriumok dominálták. Ehhez képest a BF csecsemők sokkal heterogénebbek voltak a phyla összetételük tekintetében. Három BF csecsemő mikrobiotájában az Actinobacteriumok domináltak, az egyikben a Proteobacteriumok domináltak, az egyikben a Bacteroidetes dominált, a másikban pedig a phyla nagyon egyensúlyban volt. Amint az a 2c. Ábrán látható, a külsõ FF csecsemõ mikrobiota kivételével a BF csecsemõ mikrobiota magasabb alfa-változatosságot mutatott, mint az FF csecsemõ mikrobiota, amelyet a Shannon-Wiener index számított.
Az étrend hatása a csecsemő mikrobiotájára. A BF (szoptatott) csecsemők (zöld) a filogenetikai összetétel tekintetében több heterogenitást mutattak, mint az FF (tápszeres táplálású) csecsemők (kék). a) A taxon hozzárendelés (phylum level) variabilitás a BF és FF minták esetében 16S rRNS GreenGenes-hez való igazítással (lásd Anyagok és módszerek). Az étrendcímke-permutációs teszt a ∑s | ∑iεBF pis/6 - ∑iεFF pis/6 | statisztikát használva, ahol s indexek a phylum, az iεBF és az iεFF azt jelzik, hogy az i minta BF vagy FF csecsemő, és p jelöli a kapcsolódó taxont arányban elvetette azt a nullhipotézist, miszerint a filogenetikai összetétel változékonysága nem függött össze a BF/FF státusszal, P-értéke 0,011. b) Taxon-hozzárendelések az összes sörétes olvasáshoz (nem csak a 16S rRNS-homológokhoz) a PhymmBL segítségével [17]. c) Shannon-Weiner index a BF és FF csecsemők számára, jelezve az alfa-változatosságot az egyes mintáknál.
Megállapításaink megerősítéséhez a PhymmBL-t alkalmaztuk a sörétes szekvencia-leolvasások taxonómiai osztályozásához. A PhymmBL [17] a metagenomikai adatok osztályozási megközelítése, amely interpolált Markov modelleket (IMM) és Basic Local Alignment Search Tool (BLAST) használ a taxonómiai DNS szekvenciák osztályozásához. Az olvasásokat a phyla-hoz rendeltük, amint azt a 4. és 5. kiegészítő fájl összefoglalja. Míg a 16S-alapú elemzés és az egész sörétes-olvasás-alapú elemzés között általános egyetértés volt, azonosítottunk néhány következetlenséget. Ezek megfeleltek a Koenigben talált hasonló eltéréseknek et al. [18], és valószínűleg az Actinobacteriumokból származó 16S rRNS alulreprezentációjának tudhatók be. Összességében mindkét elemzés összhangban áll egy korábbi jelentéssel, amely 3 hónapos korú csecsemőknél magas Actinobacteria és Proteobacterium szintet mutat [18].
Funkcionális elemzés
A metagenomikus adatok funkcionális elemzése. Felső panel: SEED 1. szintű kategóriák, amelyekhez az összes BF vagy az összes FF minta legalább 200 olvasást leképezett. A feltérképezett leolvasások teljes számának legalább 2% -át tesztelték a BF (szoptatott) csecsemők (zöld) és az FF (tápszerrel táplált) csecsemők (kék) közötti különbségek tekintetében. Végeztünk egy permutációs tesztet a ∑iεBF pi/6 - ∑iεFF pi/6 tesztstatisztikán, ahol az iεBF és az iεFF azt jelzi, hogy az i minta BF vagy FF csecsemő, és p a kapcsolódó taxon arányt jelöli. Az FDR korrigált q-érték a virulencia kategóriában 0,058 volt. Alsó panel: a BF és FF csecsemők közötti különbségeket a SEED 2. szintű virulencia hozzárendelésében (a SEED 1. szintű virulencia kategóriában) permutációs teszt alkalmazásával értékeltük a tesztstatisztikán ∑s | ∑iεBF pis/6 - ∑iεFF pis/6 |, ahol s indexeli a SEED 2. szintű virulencia kategóriákat, és P = 0,0140.
A gén tripla CCA analízisek kanonikus összefüggései alapján az egyes expresszált gazda géneket rangsoroltuk az empirikus többváltozós kapcsolatukhoz képest a metagenómában található gének gyakoriságával. A legígéretesebb gazda gének listájának összeállításához megvizsgáltuk azon génhármasok arányát, amelyek első kanonikus korrelációs együtthatója legalább 0,85, a második kanonikus korreláció pedig legalább 0,5 volt. Ezeket választották, hogy kiemeljék az immunitás és a védekezés génkészletének északkeleti negyedében megfigyelt első és második kanonikus korrelációs pontszám gazdagodását (4a. Ábra). Az eredményül kapott immunitás- és védekező gének, a bélbiológiai gének és a példa véletlenszerű génkészletét az 5. ábra mutatja (további példa és a véletlen génkészletek reprezentatív diagramjai a 7. kiegészítő fájlban találhatók). A metagenómából származó virulencia változókkal a legerősebb empirikus többváltozós kapcsolatot mutató gének az immunitás és a védekezés génkészletéből származnak.
A gazda gének háromszoros megjelenésének gyakorisága. A génhármasok csoportjait akkor vettük fel, amikor az első kanonikus korreláció legalább 0,85, a második kanonikus korreláció pedig legalább 0,65 volt. Ezeket a szinteket önkényesen választottuk, hogy erős, többváltozós szerkezetet képviseljenek, amelyet a CCA azonosított. A géneket a legjobban teljesítő hármasok gyakorisága alapján rangsorolták. Ez kvalitatív profilt adott azoknak a géneknek a kiválasztására, amelyek empirikusan mutatják a legerősebb kapcsolatot a mikrobiom virulenciajellemzőivel. (a, b) Az immunitáshoz és a védekezéshez kapcsolódó gének messze felülmúlják a többi funkcionális kategóriát. Például a két legjobban teljesítő bélbiológiai gént valójában immun- és védekező génekként is felsorolták. c) Ezzel szemben a véletlenszerűen kiválasztott gének nem mutattak semmilyen erős többváltozós szerkezetet a mikrobiom virulenciajellemzői szempontjából.
A 11 legígéretesebb azonosított gazda-gént, funkcionális annotációjukkal és a kapcsolódó biológiai válaszokkal együtt soroljuk fel; VAV2 (angiogenezis), ALOX5 (gyulladásos válasz), SP2 (transzkripciós faktor), BPIL1 (bakteriocid), DUOX2 (peroxidáz generáció), KLRF1 (citotoxicitás), IL1A (gyulladásos válasz); AOC3 (vaszkuláris tapadás), NDST1 (gyulladás és nyálkahártya védekezése), REL (bélproliferáció és apoptotikus homeosztázis) és TACR1 (bélmozgás). Mint látható, e gének többsége immunválaszhoz kapcsolódik. A BF és az FF csecsemők 3 hónapos táplálkozási periódust követő relatív génexpressziós szintjét a 2. táblázat mutatja. Mivel a kanonikus összefüggések hármasokhoz vannak rendelve, nem pedig egyetlen génekhez, megvizsgáltuk azt is, hogy mely gének együtt mutatták a legígéretesebb többváltozós kapcsolatot a mikrobiómával változók. A 6. ábra bemutatja, hogy mely géneknél volt leggyakrabban a legjobb kanonikus összefüggés (csomópont mérete), és melyik génpár együtt ugyanazon hármasban volt a leggyakrabban a legjobb kanonikus összefüggés (az él nagysága). Ez a megjelenítés áttekintést nyújt a gének közötti szinergikus erősségről a többváltozós mikrobiom kapcsolati jellemzőinek javítása tekintetében.
Vita
A metagenomikai adatok kezdeti vizsgálatával megállapítottuk, hogy a mikrobiom „antibiotikumokkal és mérgező vegyületekkel szembeni rezisztenciája”, „III. Típusú, IV. Típusú, ESAT szekréciós rendszerek”, „vashiányozó mechanizmusok” és „virulencia” jellemzői bizonyították a differenciál érzékenységet az anyatejhez képest a tápszerhez képest. Konkrétan, míg más mikrobiomajellemzők nem szolgáltattak erős bizonyítékot a BF és FF csecsemők közötti differenciálódásra a többszörös tesztelés céljából történő igazítás után, a virulenciával kapcsolatos mikrobiális gének továbbra is erősek maradtak. Ezért transzkripptikus elemzéseinket a gazda immunitására és a védekezéshez kapcsolódó génekre összpontosítottuk. Továbbá, mivel a fejlődő emberi bél tanulmányozását végeztük, a bélbiológiában ismert géneket is megvizsgáltuk. Kezdeti differenciál expressziós elemzésünk arra utalt, hogy előzetes ismereteink génkészletei a releváns génkészleteket célozták meg.
Ezután a metagenomikus és a transzkriptomikus adatsorokat analitikusan egyesítettük egy többváltozós elemzésben, amely lehetővé tette számunkra, hogy felmérjük a mikrobiom virulencia-változói és a vizsgált béltranszkriptóm-gének közötti többváltozós kapcsolat erősségét. A vizsgált legjobban teljesítő gének rangsorolása azt mutatta, hogy a legerősebb többváltozós kapcsolat a virulencia jellemzőivel az immunitás és a védekezés génjei voltak. E megállapítás hitelességét a eleve az a várakozás, hogy a mikrobiom virulenciajellemzőivel a legerősebb kapcsolat az immunitás és a védekező gének, valamint az 1000 véletlenszerű génkészlet összehasonlítása, amelyhez az immunitás és a védekező génkészlet a transzkriptóm mértékéhez viszonyítva a 96,9. mikrobioma többváltozós szilárdság. Bár a bélbiológia kimutatott néhány erős, többváltozós kapcsolatot a mikrobiom virulencia jellemzőivel, ezek nem voltak szokatlanok a véletlenszerű génkészletekhez képest, és természetesen nem voltak olyan erősek, mint az immunitás és a védekezés génjei (4. ábra).
Következtetés
Anyagok és metódusok
Etikai nyilatkozat és tantárgyfelvétel
A kísérleti emberi protokollt az Illinoisi Egyetem és a Texas A&M Egyetemi Intézményi Felülvizsgálati Testülete hagyta jóvá, és a szülőktől a vizsgálatban való részvétel előtt tájékozott beleegyezést kaptak. A tanulmányi felvételi kritériumok és protokollok részleteit korábban leírták [12]. Egészséges, teljes időtartamú csecsemők, akik kizárólag szoptattak vagy kereskedelmi forgalomban kapható anyatej-helyettesítő tápszereket tápláltak (Enfamil LIPIL, Mead Johnson Nutrition, Evansville, IN, USA), és orvosilag igazoltan egészségesek voltak alkalmasak a vizsgálatba. A vizsgálatban részt vevő minden csecsemőről a székletmintákat a születés után három hónappal gyűjtötték.
A széklet DNS izolálása
A genomi DNS-t Yu és Morrison módszerének módosításával extrahálták [40]. A protokolltól való eltérés magában foglalta az üvegverő lépéseinek alkalmazását. Röviden: 250 mg (nedves tömeg) székletet lemértünk egy 2,0 ml-es csőbe, amely E üvegmátrixot tartalmazott (MP Biomedicals, Solon, OH, USA). Lízispuffert (1 ml; 500 mM NaCl, 50 mM Tris-HCl, 50 mM EDTA, 4% nátrium-dodecil-szulfát) adunk a csőbe, és 30 másodpercig rázzuk. A mintákat ezután 70 ° C-on 15 percig inkubáltuk. Centrifugálás után 16 000-nél g 5 percig a felülúszókat 2,0 ml-es csövekbe gyűjtöttük. Ezután 300 ul lízispuffert adunk hozzá, és a fenti lépéseket megismételjük. A felülúszóban lévő nukleinsavakat egymás után ammónium-acetáttal és izopropanollal kicsapjuk, és TE pufferben oldjuk. A kicsapódott nukleinsavakat ezután DNáz-mentes RNázzal, proteináz K-vel kezeltük, és tovább tisztítottuk QIAamp spin-oszlopon QIAgen DNS Mini székletkészletből (Qiagen, Valencia, Kalifornia, USA). A DNS minőségét 1% -os agaróz géleken ellenőriztük, majd etidium-bromid festést végeztünk. Mintánként három-négy extrakció DNS-ét egyesítettük, és koncentrációját NanoDrop 1000 spektrofotométerrel (NanoDrop Technologies, Wilmington DE, USA) számszerűsítettük.
A bélmikrobiomák szekvenálása
A székletmintákból származó DNS-t az illinoisi egyetem Keck Center for Comparative and Functional Genomics nagy teljesítményű szekvenáló és genotipizáló egységéhez juttatták el. A mintákat egy 454 Life Sciences Genome Sequencer FLX alkalmazásával szekvenáltuk GS FLX Titanium sorozatú reagensekkel (Roche, Nutly, NJ, USA). Röviden, a DNS-t frakcionáltuk (500-800 bp-os fragmensek) és csiszoltuk. Ezt követően az A és B adaptereket tartalmazó vonalkódokat Roche utasításai szerint a végekhez ligáltuk. A B adapter egy 5'-biotin-taget tartalmazott a DNS-könyvtár streptavidin gyöngyökön történő immobilizálására. A nickjavítás után a nem biotinilezett szálat felszabadították és egyszálú templát DNS (sstDNS) könyvtárként használták. A könyvtár mennyiségét Qubit reagensekkel (Invitrogen, CA, USA) határoztuk meg, és azonos molaritásra összegyűjtöttük. Az emulziós PCR-hez a DNS optimális szintjét titrálással határoztuk meg. A gyöngyöket egy PicoTiterPlate eszközre töltöttük a puskás szekvenáláshoz. A jelfeldolgozás Roche szoftver segítségével történt.
Gazdabél mRNS transzkriptóm elemzés
Mindegyik alanyból a poli A + RNS-t izoláltuk az ürülékből az előzőekben leírtak szerint [12, 41]. Az ürülékmintákban található magas bakteriális RNS szint miatt a poli A + RNS-t izolálták annak érdekében, hogy erősen dúsított emlős RNS-populációt kapjanak [12]. Ezenkívül Agilent 2100 Bioanalyzer-t alkalmaztunk a hámlasztott sejt poli A + RNS integritásának felmérésére, és a mennyiségi meghatározást NanoDrop spektrofotométerrel végeztük. A mintákat szigorúan a CodeLink ™ Gene Expression Assay kézikönyv (Applied Microarray, Tempe, AZ, USA) szerint dolgoztuk fel, és az emberi teljes genom expressziós Bioarray segítségével elemeztük, ahogy azt korábban leírtuk [13].
Metagenomikus adatok elemzése
A Shotgun 454 olvasási adatokat a következő módon dolgozták fel előre: (i) az alacsony minőségű olvasmányokat eltávolították, ha az olvasott Phred átlagértéket
- Brazil Diet Diet Pill Shop - Globális tanulmány UK
- Az anyatej és az anyai bél mikrobiota befolyásolja az antibiotikumokkal szemben rezisztens baktériumokat a csecsemő bélében
- Alopecia Areata Esettanulmány - Elit tanulás
- A bariatrikus embolizációt értékelték a BEAT elhízási vizsgálatában - Endovaszkuláris ma
- Az étrendben szereplő árpacukor csökkenti a cukorbetegség kockázatát, tanulmányi műsorok