A zsiráf genomszekvenciája rávilágít egyedi morfológiájára és fiziológiájára
Tárgyak
Absztrakt
A zsiráf impozáns termetének és a kapcsolódó kardiovaszkuláris adaptációk eredete nem ismert. Az Okapi, amelyből hiányoznak ezek az egyedülálló tulajdonságok, a zsiráf legközelebbi rokona, és hasznos összehasonlítást nyújt a zsiráf hosszú nyakának és szív- és érrendszerének mögöttes genetikai variációk azonosítására. A zsiráf és az okapi genomját szekvenálták, és összehasonlító elemzések révén azonosították azokat a géneket és útvonalakat, amelyek egyedi genetikai változásokat mutatnak és valószínűleg hozzájárulnak a zsiráf egyedi jellemzőihez. Ezen gének egy része a HOX, a NOTCH és az FGF jelátviteli utakban található, amelyek mind a csontváz, mind a szív- és érrendszeri fejlődést szabályozzák, ami arra utal, hogy a zsiráf testalkata és kardiovaszkuláris adaptációi párhuzamosan fejlődtek kis számú gén változása révén. A mitokondriális anyagcsere és az illékony zsírsavak transzportgénjei evolúciós szempontból is eltérnek a zsiráftól, és összefüggésben lehetnek annak szokatlan étrendjével, amely mérgező növényeket tartalmaz. Váratlanul jelentős evolúciós változások történtek a zsiráfban és az okapi-ban a kettős szálú törés javításában és a centroszóma funkcióiban.
Bevezetés
Okapi (Okapia johnstoni), a zsiráf legközelebbi rokona és az egyetlen fennmaradt tagja a Giraffidae család, hasznos összehasonlítást nyújt, mert nem osztja meg ezeket a zsiráf 13-ban látható egyedi tulajdonságokat. A zsiráf kilenc alfajait azonosították, amelyek a szőrzet színével és mintájával megkülönböztethetők, és reproduktív módon izolálódtak, amíg 2 mya (14., 15. hivatkozás). Két zsiráf alfaj majdnem kihalt, és a zsiráfok száma 2000 óta 40% -kal csökkent az orvvadászat és az élőhelyek elvesztése miatt 16. Mivel minden zsiráf alfajnak megvan a zsiráf nemzetség egyedi anatómiai és fiziológiai adaptációja, fontos keresztellenőrzést nyújtanak a genetikai variáció egyedi mintázatai tekintetében.
Itt szekvenáltuk a maszáj zsiráf és az okapi genomjait, és összehasonlító elemzéssel más eutheri emlősökkel 70 gént azonosítottunk, amelyek az adaptáció (MSA) többféle jeleit mutatják a zsiráfban. Ezen gének közül több a csontváz, a szív- és érrendszeri és az idegi fejlődés jól ismert szabályozóit kódolja, és valószínűleg hozzájárul a zsiráf egyedi jellemzőihez.
Eredmények
Genomszekvenálás és de novo összeszerelés
Összehasonlító genomelemzés
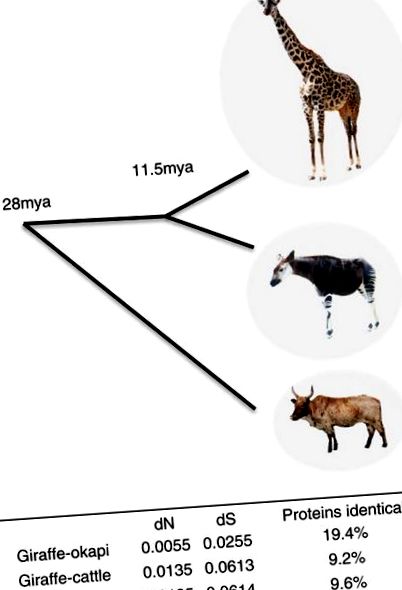
Ezen egyedi morfológiai és fiziológiai adaptációk mögött álló változások azonosításához elemeztük a zsiráf, az okapi és a szarvasmarha ortológ génjeinek kódoló szekvenciáit. A zsiráf és az okapi gének összességében nagyon hasonlóak, a fehérjék 19,4% -a azonos (1. ábra). A zsiráf és az okapi gének ugyanolyan távoli rokonságban vannak a szarvasmarhákkal, ami arra utal, hogy a zsiráf egyedi tulajdonságai nem az evolúció összességében gyorsabb következményei. A zsiráf és az okapi eltérését, a szinonim szubsztitúciók relatív arányai alapján, egy közös őstől 11,5 mya-ra becsülik (1. ábra), ami lényegesen kisebb, mint a korábbi 16 mya-os becslés (19., 20. hivatkozás). mitokondriális DNS szekvencia összehasonlítások alapján.
A zsiráf, az okapi és a szarvasmarha közötti átlagos páros szinonim szubsztitúciós divergencia (dS) becsléseket felhasználva, amelyet a pecoran közös ős (27,6 mya) kalibrált, a zsiráf és az okapi eltérése a közös őstől 11,5 mya. Okapi kép Raul654 fényképéből adaptálva.
A zsiráf adaptív evolúciója
Hetven olyan gént azonosítottak, amelyek MSA-kat mutattak ki az aminosav-szekvencia divergenciáján alapulva, amelyet emlősök ortológ fehérjéinek szomszédos csatlakozású filogenetikai elemzésével, nem szinonim szubsztitúciók dúsításával, egyedi aminosav szubsztitúciókkal emlősökben egyébként rögzített helyeken értékeltek, szubsztitúciók funkcionális változásokat okoznak a Polyphen2 által elemzés és szubsztitúciók pozitív szelekció alatt. Klaszteranalízist végeztünk 70 zsiráf MSA génkészleten, a GO Biological Process alapján, a Cytoscape 3.0 alkalmazásával (68. hivatkozás).
A csontváz növekedésének és differenciálódásának szabályozóinak alakulása
A Giraffe FOLR1 kivételesen erős bizonyítékot mutat az adaptív evolúcióra, beleértve hat pozitívan kiválasztott aminosav-szubsztitúciót, amelyek közül kettő várhatóan jelentős változást okoz a funkcióban (3b. Ábra). FOLR1 a mutációk embrióilag halálosak az egerekben 28, és hipomielinizációt és neurológiai hibákat okoznak az emberekben 29. A sejtek foláttranszportjában betöltött szerepe mellett a FOLR1 internalizálódik, feldolgozódik és a sejtmagba szállítja, ahol szabályozza az FGF és a NOTCH útvonalának elemeit 30. A FIRR1 zsiráf ezen változásai együttesen működhetnek a FGFRL1 és JAG1, az FGF és a NOTCH útvonal komponensei a főbb fejlődési adaptációk előteremtésére.
Kardiovaszkuláris és metabolikus génfejlődés
A zsiráf szív- és érrendszer úgy van kialakítva, hogy 6 m magasságban szabályozza a vérnyomást és fenntartsa az agy és a szív relatív helyzetének gyors változásával járó kardiovaszkuláris homeosztázist. A zsiráf vérnyomása 2,5-szer magasabb, mint az emberé, a szív bal kamrája megnagyobbodott, az alsó végtagok érfalai pedig nagyon megvastagodtak 1,31. A zsiráf bizonyítja a vérnyomást vagy a kardiovaszkuláris funkciót szabályozó nyolc gén adaptív evolúcióját, köztük a két fő adrenerg receptor α1 és β-2, az urotenzin-2b és az angiotenzin-konvertáló enzimet (4. kiegészítő táblázat). A BORG1 és az RCAN3, amelyek nagyon expresszálódnak a szívben, és állítólag fontos funkcióik vannak a sejt alakjával és a szívizom összehúzódásával kapcsolatban, szintén szignifikánsan eltérnek a zsiráf 32,33-tól. Ezen gének megfigyelt megkülönböztető változásai nyomokat adhatnak a zsiráf magas vérnyomásának, a megnövekedett szívteljesítménynek és a módosult érrendszernek az evolúciós eredetéről.
A zsiráf magas testalkata lehetővé teszi, hogy akáclevelekkel és magvakkal táplálkozzon, amelyek nagyon táplálóak, de mérgező alkaloidokat is tartalmaznak. A többi kérődzőhöz hasonlóan a zsiráfok bélmikrobái is erjesztik a növényeket, hogy olyan illékony zsírsavakat állítsanak elő, amelyek a bélhámon keresztül szállíthatók és a fő energiaforrásként szolgálnak 34,35. A zsiráf MSA-génjei közé tartoznak azok, amelyek részt vesznek az illékony zsírsavak, például a butirát (MCT1, ACSM3 és ACADS) katabolizmusában vagy az ATP-t (NDUB2 és SDHB) generáló downstream oxidatív foszforilációban (3c. Ábra). Ezenkívül ezek a fehérjék elengedhetetlenek a laktát transzportjához és anyagcseréjéhez, ami különösen fontos a szív- és érrendszeri funkciók szempontjából 36 .
A DNS és a kromoszóma-javító gének evolúciós változásai
Vita
A zsiráf fejlődési és fiziológiai szabályozó génjei, amelyek adaptív evolúciót mutatnak, gazdagodnak csontváz-, kardiovaszkuláris és idegi funkciókban. Felsorolják azokat az MSA-géneket, amelyekről nem ismert, hogy összefüggenek a csontváz, a szív- és érrendszeri vagy az idegi fejlődés szabályozásával (jobb oldali doboz).
A zsiráfban MSA-t mutató 70 gén között, FGFRL1 a legerősebb jelölt arra, hogy közvetlenül befolyásolja az axiális és a vakbélváz, valamint a szív- és érrendszer egyedi növekedését. Az FGFRL1 köztudottan nélkülözhetetlen a normális csontváz- és kardiovaszkuláris fejlődéshez emberekben és 25,26,27 egerekben, és az FGF útvonal szabályozza az 51-es somit méretét. Más génekre van szükség a nyaki csigolyák és a lábak differenciális növekedésének korlátozásához, és valószínűleg a homeotikus gének játszanak szerepet ebben a test különböző régióinak azonosságában. Három homeobox gént azonosítottunk -HOXB3, CDX4 és NEM- amelyek jelentős eltéréseket mutatnak a zsiráfban más emlősökhöz képest. A génszerkesztési módszerek megjelenése lehetőséget nyújt e hipotézisek tesztelésére a zsiráfban látható egyedi aminosav-szubsztitúciók bevezetésével a modell organizmusok homológ génjeibe és a funkcionális következmények meghatározásával. Az emlősök közül a zsiráf a legmagasabb szintű fiziológiai és szerkezeti problémákkal jár, amelyet toronymagassága okoz. Ezeknek a kihívásoknak a megoldásai, különösen a turbófeltöltéssel ellátott keringési rendszerrel kapcsolatban, tanulságosak lehetnek az emberek szív- és érrendszeri betegségeinek és magas vérnyomásának kezelésében.
Mód
Genomszekvenálás
Az Illumina TruSeq DNS PCR-mentes könyvtári előkészítő készletét két nőstény maszáj zsiráf (G.c. tippelskirchi) a kenyai MA1-ből és a Nashville Zoo-ból (NZOO), valamint egy magzati hím okapi (O. johnstoni) a White Oak Holdings-tól. A könyvtárakat a gyártó protokollja szerint készítettük 2 μg bemenet és az 550 bp méretű betétméret munkafolyamat felhasználásával. A Nextera Mate Pair mintakészítő készletet használtuk fel párpárkönyvtárak készítésére ugyanazon három mintából, a gyártó 4–8 kb méretű válogatású „Gel Plus” protokolljával. A könyvtárakat egy Illumina HiSeq 2500-on szekvenáltuk Rapid Run módban, 2 × 150 bp páros végű szekvenálással. Az összes könyvtárat a Penn State Genomics Core Facility készítette és szekvenálta a University Park-ban (PA). Specifikus gének célzott szekvenálása Rothschild-ban (G.c. rothschildi) és a netes (G.c. retulata) zsiráf olyan genomi DNS-t használt, amelyet izoláltunk a San Diego Állatkert Természetvédelmi Kutatóintézetének dr. Oliver Rydertől kapott primer fibroblaszt sejtkultúrákból.
Minőségellenőrzés és genom lefedettség
A fajok közötti variáns nukleotidokat az alábbiak szerint azonosítottuk. Azokat a szekvenciákat, amelyek a fentiekben leírtak szerint illeszkedtek a referenciagenomhoz, a referenciagenomhoz való illesztés kezdő helye szerint rendeztük. Ezeket aztán referencia alapú megközelítéssel 52 állították össze, és az összeszereléshez a régió legalább kétszeres és legfeljebb 80-szoros lefedettségét kellett figyelembe venni. Az okapi minták szekvenciáit a BWA 53 0.5.9 változatának felhasználásával a zsiráf konszenzus szekvenciához igazítottuk, alapértelmezett argumentumokkal, majd a zsiráf és az okapi közötti különbségeket a SAMtools 54 0.1.19 verziójának alapértelmezett argumentumokkal és az mpileup parancs segítségével azonosítottuk. Házon belüli szkripteket (kérésre rendelkezésre állnak) használtak a változatok helyzetének meghatározásához a (tehén vagy kutya) referencia szekvenciához képest.
Az elolvasásokat elvetettük, ha a fenti folyamat három kritérium alkalmazásával bizonyította az elégtelen olvasási minőséget vagy a genomi régió instabilitását. Először az olvasásokra volt szükség a legjobb illesztéshez a referencia-összeállításhoz, legalább 3% -kal több azonos nukleotiddal, mint a második legjobb illesztés. Másodszor, a referencia folytatásokat figyelmen kívül hagyták, ha a lefedettség mélysége túl magas vagy túl alacsony volt a Lander – Waterman statisztika szerint. Harmadszor, figyelmen kívül hagyták azokat a régiókat, amelyeknél a fajok közötti különbségek szokatlanul magasak, a duplikációk és az alacsony összetettségű régiók hatásának csökkentése érdekében. A kutya referencia-összeállításával azonosított és a későbbi elemzések során alkalmazott nukleotid-különbségek átlagos olvasási mélysége 20,0 volt az MA1-es zsiráf, 21,6 a Nashville Zoo (NZOO) zsiráf és 16,8 az okapi esetében.
Körülbelül 300 olyan gén, amely viszonylag magas dN/dS arányt mutatott a zsiráfban a tehénhez és az okapi-hez képest, hiányzott a teljes lefedettséghez képest a szarvasmarhákhoz vagy más emlősök más ortológusaihoz képest. A legtöbb esetben e gének hiányos lefedettsége annak a ténynek volt köszönhető, hogy az alkalmazott szarvasmarha-referencia-génmodell hiányos volt más emlősökhöz képest. Ezeknek a géneknek a megjegyzésének kiegészítése érdekében azonosították az ezeket a géneket tartalmazó zsiráf és okapi állványokat. A megfelelő állványokat a Genewise 55 annotációs program segítségével szarvasmarháktól vagy emberektől származó teljes referencia kódoló szekvenciák alkalmazásával elemeztük. A legmagasabb szintű bizalommal és információval rendelkező Ensembl referencia-átiratokat (TSL: 1, GENECODE basic, APPRIS P1) használtuk.
De novo összeszerelés
Először a párpár adataiból a TruSeq adaptereket eltávolítottuk a Nesoni alapértelmezett paramétereivel (v0.115) (https://github.com/Victorian-Bioinformatics-Consortium/nesoni). Ezután a KmerGenie (v1.6269) 56 alapértelmezett paraméterekkel lett végrehajtva mindkét adathalmazon, hogy meghatározzuk a legjobb k-mer méreteket az összeszereléshez. Az állványokat a SOAPdenovo2 (v2.04) 57 alkalmazásával állították össze, a k-mer méretét 91-re állítva a zsiráf-adatkészletre és 81-et az okapi adatkészletre, és engedélyezve az ismételt felbontást (-R paraméter). Végül az állványok hiányosságait kitöltötte az alapértelmezett paraméterekkel rendelkező GapCloser (v1.12) segítségével.
Ugyanazokat a páros végű és páros olvasmányokat, amelyeket az összeszereléshez használtak, visszaképeztük a zsiráf és az okapi összeállításra. A BWA-MEM programot alapértelmezett paraméterekkel hajtották végre, és a statisztikákat a „samtools stats” eszközzel vonták ki. Figyelemre méltó, hogy a megfelelően feltérképezett párpárok százalékos aránya alacsonyabb volt, mint a párosított végeknél, mivel a párpár nagyobb átmérője nagyobb valószínűséggel térképezi fel a különböző állványokat.
Igazítások és génfák
A szekvenciák összehangolása előtt minden szekvencián tblastn-t futtattunk a megfelelő tehénfehérje RefSeq szekvenciával (letöltve az Ensembl-től). Ez biztosította a képeltolás-indelek korrekcióját, mivel megjegyezték, hogy egyes szekvenciák huzatminőségűek, és lehetnek szekvenciahibák. A szekvenciákat a MUSCLE 3.8 kiadás (58. hivatkozás) segítségével igazítottuk, és a filogenetikai fákat a PhyML 3.0 verzióval (59. hivatkozás) készítettük. A PhyML valószínûségalapú fa keresõ algoritmust használ az optimális filogenitás megtalálásához. Bootstrapping (n= 100) használtuk a kapott filogenikák robusztusságának tesztelésére.
Pozitív szelekciós elemzések
A nukleotid- és aminosav-szubsztitúciók értékelése
További információ
Csatlakozási kódok: A szekvencia adatai G. camelopardalis tippelskirchi (MA1 és NZOO) és O. johnstoni (WOAK) a Short Read Archive-ban kerültek letétbe SRP071593 projektszámmal (BioProject PRJNA313910) és az NZOO: SRX1624609 és MA1: SRX1624612 csatlakozási kódokkal. Az Egész Genom Sörétes projekt G. camelopardalis tippelskirchi (MA1) a DDBJ/ENA/GenBanknál letétbe helyezték az LVKQ00000000 csatlakozási cím alatt, és az ebben a cikkben leírt verzió az LVCL01000000 verzió. Az Egész Genom Sörétes projekt O. johnstoni (WOAK) letétbe helyezték a DDBJ/ENA/GenBanknál az LVCL00000000 csatlakozás alapján, és az ebben a cikkben leírt verzió az LVCL01000000 verzió.
Hogyan idézhetem ezt a cikket: Agaba, M. et al. A zsiráf genomszekvenciája rávilágít egyedi morfológiájára és fiziológiájára. Nat. Commun. 7: 11519 doi: 10.1038/ncomms11519 (2016).
- FDA Fogyás Fórum Fogyás Az Impact Genome Project®
- Egyél kevesebbet, többet mozogj ’- a fogyás egyszerű a Second Nature Guide-ban
- A bél-mikroba csere segít az egereknek a súly elhullásában a Nature News; Megjegyzés
- Fedezze fel, mit árul el teste alakja az egészségéről 20 Fit
- DIY természetes illóolaj hajszérum; Természet; s Lab