Diéta és az emberi amiláz gén kópiaszám-változásának alakulása
George H. Perry
1 School of Human Evolution and Social Change, Arizona State University, Tempe, AZ 85287, USA
2 Patológiai Osztály, Brigham és Női Kórház, Boston, MA 02115, USA
Nathaniel J. Dominy
3 Antropológiai Tanszék, Kaliforniai Egyetem, Santa Cruz, CA 95064, USA
Katrina G. Claw
1 School of Human Evolution and Social Change, Arizona State University, Tempe, AZ 85287, USA
4 Élettudományi Kar, Arizona Állami Egyetem, Tempe, AZ 85287, USA
Arthur S. Lee
2 Patológiai Osztály, Brigham és Női Kórház, Boston, MA 02115, USA
Heike Fiegler
5 The Wellcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Egyesült Királyság
Richard Redon
5 The Wellcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Egyesült Királyság
John Werner
4 Élettudományi Kar, Arizona Állami Egyetem, Tempe, AZ 85287, USA
Fernando A. Villanea
3 Antropológiai Tanszék, Kaliforniai Egyetem, Santa Cruz, CA 95064, USA
Joanna L. Mountain
6 Antropológiai Tudományok Tanszék, Stanford Egyetem, Stanford, CA 94305, USA
Rajeev Misra
4 Élettudományi Kar, Arizona Állami Egyetem, Tempe, AZ 85287, USA
Nigel P. Carter
5 The Wellcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Egyesült Királyság
Charles Lee
2 Patológiai Osztály, Brigham és Női Kórház, Boston, MA 02115, USA
7 Harvard Medical School, Boston, MA 02115, USA
Anne C. Stone
1 School of Human Evolution and Social Change, Arizona State University, Tempe, AZ 85287, USA
Társított adatok
Absztrakt
A keményítőfogyasztás kiemelkedő jellemzője a szárazföldi környezetben élő mezőgazdasági társaságoknak és vadászó-gyűjtögetőknek. Ezzel szemben az esőerdők és a sarkkörös vadászó-gyűjtögetők, valamint néhány pásztorember sokkal kevesebb keményítőt fogyaszt. Ez a viselkedési variáció felveti annak lehetőségét, hogy az amilázra, a keményítő hidrolíziséért felelős enzimre 4 különböző szelektív nyomás hatott. Megállapítottuk, hogy a nyál amiláz gén (AMY1) kópiaszáma pozitívan korrelál a nyál amiláz fehérje szintjével, és hogy a magas keményítőtartalmú étrendű populációkból származó egyének átlagosan több AMY1 kópia van, mint a hagyományosan alacsony keményítőtartalmú étrendben. E populációk egy részhalmazában található más lokuszokkal való összehasonlítások arra utalnak, hogy az AMY1 kópiaszám differenciálódás szintje szokatlan. Ez a példa a pozitív szelekcióra egy kópiaszámú változó génen az első az egyik az emberi genomban. A magasabb AMY1 kópiaszám és fehérjeszint valószínűleg javítja a keményítőtartalmú ételek emésztését, és pufferolhat a bélbetegség fitneszcsökkentő hatásaival szemben.
A hominin evolúciót jelentős étrendi elmozdulások jellemzik, amelyeket részben a kőeszközök technológiájának fejlesztése, a tűz elleni védekezés, valamint legutóbb a növények és állatok háziasítása indított el 5 - 7. A keményítő például az emberi étrend egyre hangsúlyosabb elemévé vált, különösen a mezőgazdasági társadalmak körében 8. Ezért ésszerű, hogy az amiláz emberekben és közeli főemlős rokonainkban történő evolúciójának tanulmányozása betekintést nyújthat ökológiai történelmünkbe. Mivel az emberi nyálamiláz gén (AMY1) a 9., 10. példányszámban nagy eltéréseket mutat, először azt értékeljük, hogy van-e funkcionális összefüggés az AMY1 kópiaszám és a nyálban lévő amiláz fehérje expresszió szintje között. Ezután meghatározzuk, hogy az AMY1 kópiaszám eltér-e a modern emberi populációk között az étrendi keményítő kontrasztos szintjétől.
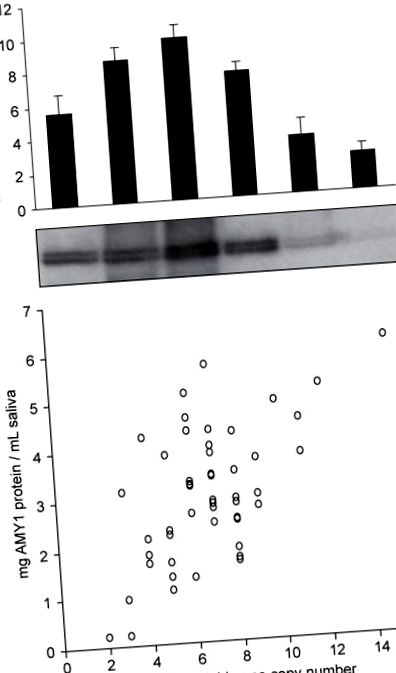
Az AMY1 kópiaszám variáció és a nyál amiláz fehérje expressziója. (a,b) Ugyanezektől az amerikai amerikai személyektől becsültük a diploid AMY1 gén kópiaszámot qPCR-rel (a) és az amiláz fehérje szintje a nyálban Western blot (b). A hibasávok jelzik az s.d. (c) Kapcsolat az AMY1 diploid kópiaszám és a nyál amiláz fehérje szintje között (n = 50 európai-amerikai). Az AMY1 fehérje szintjének jelentős mértékű variációját nem magyarázza a kópiaszám (R 2 = 0,351), amely tükrözheti az AMY1 expressziójára gyakorolt egyéb genetikai hatásokat, például a regulációs régió egy nukleotid polimorfizmusát (SNP) vagy nem genetikai tényezőt, amely magában foglalhat egyedi hidratációs állapot, stresszszint és rövid távú étkezési szokások.
Diéta és AMY1 példányszám-variáció. (a) A qPCR-re becsült AMY1 diploid kópiaszám-gyakorisági eloszlások összehasonlítása a hagyományos étrenddel rendelkező populációk esetében, amelyek sok keményítőben gazdag ételt tartalmaznak (magas keményítőtartalmú), valamint a hagyományos étrenddel rendelkező populációk esetében, amelyekben alig vagy egyáltalán nincs keményítő (alacsony keményítőtartalmú). (b) A diploid AMY1 kópiaszám kumulatív eloszlási diagramja a vizsgálat mind a hét populációja esetében.
Az AMY1 példányszám-becslések nagyfelbontású szálas FISH-hitelesítése. A vörös (~ 10 kb) és a zöld (~ 8 kb) próbák felölelik a teljes AMY1 gént, és egy retrotranszpozont közvetlenül az AMY1 előtt (és egyedülállóan). (a) A japán GM18972 egyedet qPCR becslése szerint 14 (13,73 ± 0,93) diploid AMY1 gén kópia jellemzi, összhangban a rostos FISH-eredménnyel, az egyik allél 10, a másik négy példányban. (b) A qPCR szerint Biaka GM10472 egyednek 6 (6,11 ± 0,17) diploid AMY1 gén kópia van, összhangban a rost FISH eredményeivel. (c) Megerősítették, hogy a csimpánz referencia egyén (Clint; S006006) két diploid AMY1 gén kópiával rendelkezik.
Az AMY1 kópiaszám-variáció népességi mintái nem felelnek meg az elvárásoknak a genetikai sodródás egyszerű regionális alapú modellje alapján: magas és alacsony keményítőtartalmú mintáink afrikai és ázsiai populációkat egyaránt tartalmaznak, ami arra utal, hogy az étrend erősebben jósolja az AMY1 kópiaszámot, mint földrajzi közelség. E megfigyelés alapján feltételeztük, hogy a természetes szelekció befolyásolhatta az AMY1 kópiaszámot bizonyos emberi populációkban. Egy ilyen hipotézist azonban kizárólag qPCR eredményeink alapján nem tudunk szigorúan tesztelni, részben azért, mert hiányoznak más lokuszok összehasonlító adatai. Ezért a következő lépésben tömb alapú összehasonlító genomi hibridizációt (aCGH) hajtottunk végre a jakut populáció mintáján egy Whole Genome TilePath (WGTP) tömb platformmal, amelyet Redon és munkatársai 11 korábban használtak a kópiaszám variációjának genomszintű mintáinak leírására 270-ben. egyének (a HapMap gyűjtemény), beleértve ugyanazt a japán populációs mintát, mint a tanulmányunkban. A Yakut aCGH kísérletekhez ugyanazt a referencia DNS-mintát (NA10851) használtuk, mint az előző vizsgálatban 11, megkönnyítve a japán és jakut relatív intenzitás log2 arányának összehasonlítását a tömb 26 574 bakteriális mesterséges kromoszóma (BAC) klónján, köztük két klónon feltérképezve az AMY1 lokuszra.
Japán-jakut másolatszám-differenciálás az AMY1-en, összehasonlítva más, az egész genomra kiterjedő lókuszokkal. (a) A WGTP aCGH relatív intenzitás log2 arányainak frekvenciaeloszlása az AMY1-leképezett Chr1tp-6D2 klónból japán és jakut egyének számára. (b) A japán és a jakut közötti átlag log2 arány minden autoszomális WGTP-klón esetében, amelyek kópiaszám változó volt mindkét populációban. A Chr1tp-6D2 és a Chr1tp-30C7 AMY1-leképezett klónokat folytonos vörös és kék körökként ábrázolják.
Összefoglalva, megmutattuk, hogy az emberi AMY1 gén kópiaszámának változása összhangban van az étrenddel összefüggő szelekciós nyomásokkal, ami bizonyítja a keményítőtartalmú ételek fontosságát az emberi evolúcióban. Míg az amiláz lokusz az egyik legváltozóbb az emberi genomban a 10. példányszám tekintetében, egyáltalán nem egyedi; egy nemrégiben végzett, egész genomra kiterjedő felmérés 1447 kópiaszám-változó régiót azonosított 270 fenotípusosan normális emberi egyén között 11, és valószínűleg még sok ilyen régiót fognak felfedezni a kópiaszám-variáció detektálási technológia fejlődésével. Ésszerű feltételezni, hogy az AMY1-től eltérő kópiaszám-variánsok erős természetes szelekciós nyomásnak vannak kitéve vagy voltak, különös tekintettel a transzkripciós és transzlációs szintekre gyakorolt lehetséges hatásukra (pl. 29. hivatkozás). Az emberek közötti, valamint az emberek és más főemlősök kópiaszám-variációinak jellemzése ígéretek szerint jelentős betekintést nyújt evolúciós történelmünkbe.
Mód
Minták
Bukkális tamponokat és nyálat gyűjtöttünk tájékoztatott beleegyezés alapján 50 18-30 éves európai-amerikai embertől (Arizonai Állami Egyetem IRB protokollja, 0503002355). A nyálat 3 percig gyűjtöttük a nyelv alól. Bukkális tamponokat gyűjtöttünk a Hadzától (n = 38) és a Datogtól (n = 17) Tanzániából (Stanford Egyetem IRB-protokollja: 9798-414). Biakából (Közép-afrikai Köztársaság; n = 32), Mbutiból (Kongói Demokratikus Köztársaság; n = 15) és Jakutból (Szibéria; n = 25) származó genomi DNS-ek a HGDP-CEPH Humán Genom Diverzitás Sejtpályázatából származnak. 45 japán, 4 további Biaka limfoblasztoid sejtvonalát és a csimpánz genom szekvencia donorját (Clint) a Coriell Orvosi Kutató Intézetből szereztük be. A rutin állatorvosi vizsgálatok során a teljes vért gyűjtötték a különböző állattani és kutatási létesítményekben elhelyezett csimpánzokból és bonobókból. Két további bonobo mintát nyertünk az Integrated Primate Biomaterials and Information Resource-ból. A DNS-t standard módszerekkel izoláltuk.
Számbecslés másolása
A qPCR primereit (3. kiegészítő táblázat online) úgy terveztük, hogy specifikus legyen az AMY1-re (vagyis az AMY2A és az AMY2B szekvenciájának eltérése) az emberi és a csimpánz referencia genom szekvenciák alapján. Egy korábbi tanulmány az AMY1 egyetlen (haploid) példányáról számolt be egy csimpánz 18 esetében, valamint egy friss elemzésről Cheng és mtsai. 19 nem talált bizonyítékot az AMY1 közelmúltbeli duplikációjára Clint számára. A FISH szál segítségével meggyőződtünk arról, hogy Clintnek van-e az AMY1 két diploid másolata (3c. Ábra). Ezért képesek voltunk megbecsülni a diploid kópiaszámot az emberi DNS-ek relatív AMY1 mennyisége alapján, összehasonlítva a Clint DNS-ből felépített standard görbével. A TP53 gén fragmensét is amplifikáltuk, hogy beállítsuk a DNS hígítási mennyiségének változását. A mintákat három példányban, a standardokat két példányban futtattuk. A kísérleteket a 20. leírás szerint hajtottuk végre és elemeztük .
Western blot elemzés
A fehérjemintákat úgy állítottuk elő, hogy a nyálmintákat 2% -os nátrium-dodecil-szulfátban (SDS) oldottuk és 100 ° C-on 5 percig melegítettük. Ezeket a mintákat mini SDS-poliakrilamid géleken elemeztük és polivinilidén-difluorid (PVDF) membránokra (Immobilon-Millipore) vittük át. A mennyiségi meghatározás céljából minden gélen ismert mennyiségű emberi nyál amiláz fehérje mintát (Sigma) futtattunk, minden mintához 5 μl nyállal. Transzfer után a membránokat 1,5 órán át inkubáltuk emberi nyálamiláz (Sigma) ellen termelt primer antitestekkel. A membránokat mostuk, és kecske nyúlellenes alkalikus foszfatázzal konjugált IgG szekunder antitesteket (Pierce) adtunk hozzá 1 órán át. A membránokat 5 percig ECF szubsztrátnak (Amersham Biosciences) tettük ki, és foszfor-képalkotó segítségével elemeztük. A fehérje sávok számszerűsítését ImageQuant szoftverrel (Molecular Dynamics) végeztük.
Rost HAL
A DNS-szálakat úgy állítottuk elő, hogy a tenyésztett limfoblaszt-sejteket 5 millió sejtenként 300 μl sejtlízis pufferrel (Gentra Systems) gyengén lizáltuk. 10 μl lizátumot egy poli-L-lizinnel bevont tárgylemezre (LabScientific) helyeztünk, és mechanikusan kinyújtottunk egy fedőlemez szélével. 30 másodperc elteltével 300 μl 100% -os metanolt adunk a szálak rögzítésére. A tárgylemezeket 5 percig 37 ° C-on szárítottuk, majd szobahőmérsékleten (RT) tároltuk.
PCR termékpróbákat készítettünk (i) magából a teljes AMY1 génből (~ 10 kb; piros a képeken), és (ii) a retrotranszpozonból, amely közvetlenül az összes AMY1 kópia előtt található, de nem hasnyálmirigy amiláz gének vagy amiláz pszeudogének (~ 8 kb; zöld a képeken); míg a génpróba nem feltétlenül specifikus az AMY1-re minden hibridizációs körülmény között (AMY1 szekvencia divergencia AMY2A-val és AMY2B = 7,5% és 7,1%, ill. Hosszú távot, majd beágyazott PCR-t használtunk az egyes régiókhoz (a primereket és a körülményeket a 3. kiegészítő táblázat tartalmazza online). A PCR termékeket DNS Clean és Concentrator oszlopokkal tisztítottuk (Zymo).
Minden beágyazott PCR-termékhez 750 ng-t kombináltunk 20 μl 2,5x random random primerrel (BioPrime aCGH Labeling Module, Invitrogen) 39 μl össztérfogatban, 5 percig 100 ° C-on, majd 5 percig jégen. Ezután 5 μl 10x dUTP és 1 μl Exo-Klenow töredék (BioPrime modul), vagy 5 μl (5 nmol) Biotin-16-dUTP (Roche; génszonda) vagy 5 μl (5 nmol) Digoxigenin-11-dUTP ( Roche-t (upstream próba) adtunk hozzá, és 5 órán át 37 ° C-on inkubáltuk. A megjelölt termékeket Microcon Centrifugal Filter Devices készülékkel (Millipore) tisztítottuk, háromszor 300 μl 0,1x SSC-vel mossuk, eluálva 50 μl H2O-val. Minden 1 μg jelölt DNS-hez 10 μg humán Cot-1 DNS-t (Invitrogen) adtunk.
Minden kísérlethez 500 ng jelölt DNS-t egyesített beágyazott PCR-reakciókból egyesítettünk, liofilizáltunk, 10 μl hibridizációs pufferben (50% formamid, 20% dextrán-szulfát, 2x SSC) rekonstruáltunk, és hozzáadtuk a tárgylemezhez (18 × 18 mm fedőüveg; Fisher). A szálakat és a próbákat együtt denaturáltuk (95 ° C 3 percig), és párásított kamrában (40 ° C-on 37 ° C) hibridizáltuk. A tárgylemezt 0,5x SSC-ben mossuk 75 ° C-on 5 percig, majd 3 alkalommal mossuk 1x PBS-ben szobahőmérsékleten 2 percig. Ezután a rostokat 200 μl CAS-blokkkal (Zymed) és 10% -os normál kecskeszérummal (Zymed) inkubáltuk 20 percig szobahőmérsékleten, HybriSlip (Invitrogen) alatt. Háromlépéses detektálást/amplifikációt alkalmaztunk (reagensekkel 200 μl CAS blokkban/10% normál kecskeszérumban). Mindegyik lépés 30 percig volt szobahőmérsékleten HybriSlip alatt, majd háromszor mossuk 1x PBS-ben 2 percig szobahőmérsékleten: (i) 1: 500 anti-digoxigenin-fluoreszcein, Fab-fragmensek (Roche) és 1: 500 Strepavidin, Alexa Fluor 594 konjugátum (Invitrogen); (ii) 1: 250 nyúl anti-FITC antitest (Zymed) és 1: 500 biotinilezett anti-streptavidin (Vector Laboratories); (iii) 1: 100 kecske nyúlellenes IgG-FITC (Zymed) és 1: 500 Strepavidin, Alexa Fluor 594 konjugátum. A képeket egy Olympus BX51 fluoreszcens mikroszkóppal rögzítették Applied Imaging kamerával és elemezték az Applied Imaging Genus szoftverével.
aCGH elemzés
Az aCGH kísérletekhez egy nagyméretű inszert klón DNS mikroarray-t használtunk, amely az emberi genomot lefedte a csempézési út felbontásában 30. A teszt (jakut egyedek) és a referencia (NA10851) genomi DNS mintákat Cy3-dCTP-vel és Cy5-dCTP-vel (NEN Life Science Products) jelöltük, és együtt hibridizáltuk a tömbhöz. Mindegyik minta esetében elvégeztek egy párhuzamos kísérletet festékcserével a hamis-pozitív hibaarány csökkentése érdekében. A címkézést, a hibridizációt, a mosásokat és az elemzéseket a 11., 30. leírás szerint hajtottuk végre .
- DZ Ep 22 CJ Hunt, A tökéletes emberi étrend - a megzavarási zóna
- Esszé az egészséges táplálkozásról A legjobb 7 esszé Emberi biológia
- Ciklikus keto diéta Minden, amit tudnia kell a PINKVILLA ketogén étrend ezen változatáról
- A táplálkozás alakulása az élet állatfáján keresztül - Román ‐ Palacios - 2019 - Evolution Letters -
- Fogyókúra és gének kifejezése Ön az, amit Stanford Online