Nincs bizonyíték arra, hogy Wolbachia táplálkozási társ-kötelező endoszimbiont lenne a levéltetű Pentalonia nigronervosa-ban
Alejandro Manzano-Marín
UMR 1062 Biológiai Központ a Gestion des Populations-től, INRA, CIRAD, IRD, Montpellier SupAgro, Univ. Montpellier, 755 avenue du campus Agropolis, Montpellier, Franciaország
Társított adatok
Absztrakt
Kötelező szimbiotikus asszociációk sokféle táplálékban korlátozott táplálékkal rendelkező állatban vannak. A levéltetvek (hemiptera: Aphididae) szinte univerzálisan Buchnera aphidicola baktériumokat fogadnak el a speciális szervekben (úgynevezett bakteriomák). Ezek a baktériumok ellátják a levéltetvet az étrendjükből hiányzó nélkülözhetetlen tápanyagokkal (azaz esszenciális aminosavakkal és néhány B-vitaminnal). Egyes levéltetvek nemzetségei, például a Lacninae alcsaládba tartozó fajok társ-kötelező asszociációkat alakítottak ki a másodlagos endoszimbiontokkal, amelyek a biotin- és riboflavin-bioszintetikus gének elvesztéséből származnak. Ebben a tanulmányban újra elemzem a Pentalonia nigronervosa banántetűből korábban publikált szekvenálási adatokat. Megmutatom, hogy a metabolikus következtetés De Clerck et al. (Microbiome 3:63, 2015) helytelenek, és valószínűleg nem megfelelő módszerek alkalmazásából erednek. Ezenkívül megvitatom, hogy az antibiotikum-kezelés elemzésének látszólag elfogult értelmezése, valamint a helytelen genom-alapú anyagcsere-következtetés hogyan eredményezte azt a téves felvetést, hogy „a B. aphidicola és a Wolbachia között kötelezően fennálló szimbiózis fordul elő a banántetűben”.
Fő szöveg
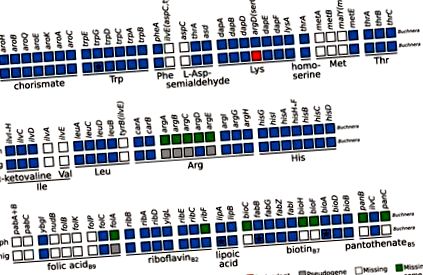
A különféle levéltetű fajok kötelező szimbiotikus konzorciumainak esszenciális aminosav- és kiválasztott B-vitamin- és ko-faktor bioszintetikus metabolikus képességei. Buchnera metabolikus képességeit összefoglaló ábra „mono-szimbiózisos” Aphidinae levéltetvekből (Aph) és P. nigronervosa (Pnig). Az „Aph” sorok több Aphidinae faj összeomlott ábrázolását jelentik (lásd az S3 kiegészítő táblázatot az 1. kiegészítő fájlban). Oszlopnévként a bioszintetikus folyamatban részt vevő enzimeket kódoló gének nevét használják. Minden sor dobozai a Buchnera genom által kódolt géneket ábrázolják. Alul a vonal alatt a név által meghatározott vegyülethez vezető útvonalban részt vevő gének aláhúzása vonalak alatt. Az aminosavak esetében hárombetűs rövidítését használják
Következtetés
Röviden, De Clerck és mtsai. [1] nem nyújtanak bizonyítékot arra, hogy a P. nigronervosa elemzett populációiban a Wolbachia látszólagos rögzülése annak köszönhető, hogy ez a szimbiont táplálkozási társkötelezettség volt ebben a levéltetvben. A szekvenálási adatok újbóli elemzése feltárta a P. nigronervosa táplálkozási társ-kötelező szimbiózisának genomon alapuló következtetéseit. Ezenkívül a primer szekvenciák vizsgálata feltárta, hogy a legtöbb primer pár nem egyezik a P. nigronervosa Buchnerában megcélzott szekvenciáival. Végül az antibiotikum-kezelés eredményeinek alternatív magyarázataik vannak, amelyeket a szerzők nem vitattak meg, és ezt nem lehet elvetni érzékenyebb elemzések nélkül. Ettől függetlenül Wolbachia látszólag következetes jelenléte ennek ellenére más folyamatokkal is magyarázható, például valamiféle reprodukciós manipulációval, vírusfertőzések elleni védelemmel vagy a mintavételezett levéltetű törzsekkel, amelyek valóban egy vagy néhány eredetileg fertőzött „szuperklónus (ok) hoz tartoznak”. szorosan kapcsolódó Wolbachia endosymbionts.
Anyagok és metódusok
Az adatgyűjtés és a genom összeállításának szekvenálása
A fa szekvenáló könyvtárak (SRR1661114, SRR1662246, SRR1662249) De Clerck és mtsai. [1] letöltötték az NCBI SRA adatbázisából (csatlakozás SRX766492) a BioProject oldalról (https://www.ncbi.nlm.nih.gov/bioproject/268300, utoljára 2019. április 15-én látogatták meg). A letöltött fastq fájlok ellenőrzése a fastQC v0.11.7 használatával (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/, utoljára 2019. április 15-én volt elérhető) feltárta az olvasások szennyeződését a TruSeq adapter indexeivel 4, 5 és 6, valamint egy ismeretlen, T + G-ben gazdag adapter („TGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGT”). Ezen adapterek kivágásához az olvasott adatkészletekből és a minőségi vágás elvégzéséhez a fastx_clipper-t használtam, majd a fastx_quality_trimmer-t, amely jobb farokminőségű vágást hajtott végre (20-as minimális minőségi küszöböt használva), és 50 bp-nél rövidebb olvasmányokat elvetett (opciók -t 20 -l 50). Ezenkívül a PRINSEQ v0.20.4 [10] -et használtuk a meghatározatlan nukleotidokat tartalmazó olvasások eltávolítására, valamint a pár nélkül maradt szűrők szétválasztására a szűrési és vágási folyamat után. A három könyvtár összeállítása a SPAdes v3.10.1 [11] alkalmazásával történt, a 33, 55 és 77 k-merekkel.
Összeállított szekvenciák Buchnerához és Wolbachiához való kapcsolása
Először minden 200 bp-nél rövidebb és 3-nál alacsonyabb átlagos k-mer lefedettségű állványt ledobtak. Ennek eredményeként 10 097-et (az állványok teljes számának 3,74% -át) eredményeztek, amelyeket azután BLASTX-alapú öblítéshez használtak fel. Ez a borsótetű fehérjék és Wolbachia és Buchnera baktériumok proteomjának adatbázisának felépítésével történt. A mitokondrium szekvenciák kimutatására külön adatbázist építettek a levéltetű Schizaphis graminun által kódolt mitokondrium fehérjékről. Mindezek a fehérjeadatok De Clerck és mtsai publikációja előtt rendelkezésre álltak. [1]. A hozzáférési számok és a törzsekkel kapcsolatos információk az S4 kiegészítő táblázatban találhatók (1. kiegészítő kék fájl). A fent említett adatbázisokkal szemben egy BLASTX keresést hajtottak végre a szűrt állványok lekérdezésként (-soft_masking true -seg yes -max_target_seqs 10000 -value 1e-3), majd az állványt a legjobb BLASTX találattal rendelkező szervezethez rendelték az egyes állványokhoz. Ezeket a tartályokat ezután leszűrtük úgy, hogy mind a Buchnerának, mind a Wolbachiának a minimális k-mer lefedettségi küszöbértékét 10-re állítottuk. Ennek eredményeként az egyes baktériumokhoz 135, illetve 1309 állványt rendeltek.
A „hiányzó” és a „táplálkozási gének” azonosítása Buchnerában
A bejelentett primerek alkalmassága De Clerck és mtsai. [1] (megtalálható az „1. kiegészítő fájlban: S1. Táblázat”) úgy ítélték meg, hogy összehangolták őket a jelen munkában azonosított és jelentett génszekvenciával az AliView v1.26 segítségével [13].
- A táplálkozási terápia új megközelítései
- Burgonya táplálkozási tényei Az egészséges táplálkozás SF kapuja
- Új bizonyíték a tesztoszteron terápia hatékony elhízáskezelés férfiaknál
- Éttermi menü címkézési politikája A bizonyítékok és ellentmondások áttekintése
- A kisérbetegség Joanna Wardlaw az Age Age bizonyítékokat vizsgálja