Az RNF4 kölcsönhatásba lép a multiSUMOylated ETV4-vel
Elisa Aguilar-Martinez
1 Biológiai, Orvostudományi és Egészségügyi Kar, Manchesteri Egyetem, Manchester, M13 9PT, Egyesült Királyság
Baoqiang Guo
1 Biológiai, Orvostudományi és Egészségügyi Kar, Manchesteri Egyetem, Manchester, M13 9PT, Egyesült Királyság
Andrew D. Sharrocks
1 Biológiai, Orvostudományi és Egészségügyi Kar, Manchesteri Egyetem, Manchester, M13 9PT, Egyesült Királyság
Az ADS megalkotta a tanulmányt. Az EAM, BG és ADS tervezték a kísérleteket. Az EAM és a BG elvégezte a kísérleteket. Az EAM, BG és ADS írta és szerkesztette a kéziratot, és beleegyezett a végleges tartalomba.
Társított adatok
2A. És C. ábra. Nyers adatok. A teljes western blotok láthatóak, és a 2. ábra paneljeibe felvett területek kiemelve vannak (dobozokkal jelölve).
2B. Ábra. Nyers adatok. A teljes western blotokat mutatjuk be, és a 2. ábra paneljeibe felvett területeket kiemeljük (négyzetekkel jelölve).
A cikkhez társított adatok a Creative Commons Zero "Nincs jog fenntartva" adatmentesség (CC0 1.0 Public Domain dedikáció) feltételei szerint érhetők el. http://creativecommons.org/publicdomain/zero/1.0/
Verzióváltozások
Felülvizsgált. Módosítások az 1. verziótól
Mindkét játékvezető értékes javaslatokat tett a cikk érthetőségének növelése érdekében. Számos szöveges változtatást és módosítást hajtottunk végre az ábrákon a felvetett kérdések kezelése érdekében. Változások az ábrákon: Két további sávot adtunk az 1C. Ábrához, amelyek azt mutatják, hogy az általunk megfigyelt multi-SUMOylation szintek elegendőek ahhoz, hogy elősegítsék az RNF4 kötődését az ETV4-hez (mivel nem figyelhető meg kötés a nem SUMOylatable ETV4 (K1234R) mutánshoz ). Egy további ábra panel (1F) került hozzá annak bemutatására, hogy a SUMO-val (K11R) módosított ETV4 (amely nem képezhet láncokat) továbbra is kötődik az RNF4-hez, és ezért a kötődést a multi-SUMO közvetíti. A molekulatömeg markereit végig adtuk hozzá. A vázlatot úgy módosítottuk, hogy megmutassa a fehérjék megfelelő számú SIM-jét és a SUMO-módosítási helyeit. Változások a szövegben: További vita került az RNF4-polySUMO kölcsönhatások strukturális vizsgálataival kapcsolatban. Ezen túlmenően megvitatják az ETV4 N-terminális régiójában bekövetkező strukturális változások SUMO bemutatásának következményeit. Számos módszertani pontosítást adtak hozzá, és nagyobb pontosságot használtak az alkalmazott különböző RNF4 és ETV4 konstrukciók leírásához. Az ábrákon látható új adatok megvitatása kiegészült.
Peer Review összefoglaló
| 2017. február 17 | Manuel S. Rodriguez | 2. verzió | Jóváhagyott |
| 2016. december 9 | Manuel S. Rodriguez és Elisa Da Silva-Ferrada | 1. verzió | Foglalásokkal jóváhagyva |
| 2016. november 28 | Tony Hunter | 1. verzió | Jóváhagyott |
Absztrakt
A protein SUMOylation fontos szabályozási esemény, amely számos fehérje aktivitását megváltoztatja. A legújabb bizonyítékok azt mutatják, hogy a polySUMO-láncok kiváltó tényezőként működhetnek az RNF4 ubiquitin-ligáz szubsztrátokhoz irányításában, hogy forgalmuk az ubiquitin-úton keresztül történjen. Az RNF4 több SUMO interakciós motívumot (SIM) használ ezekhez a láncokhoz való kötődéshez. A polySUMO láncok mellett azonban egy multimer kötőfelület, amelyet egy fehérjére vagy komplexre több maradék egyidejű SUMOilezésével hoztak létre, szintén platformot teremthet multi-SIM fehérjék, például az RNF4 toborzásához. Itt bemutatjuk, hogy a multiSUMOylated ETV4 képes kötődni az RNF4-hez, és hogy az SIM-ek egyedi kombinációjára van szükség ahhoz, hogy az RNF4 kölcsönhatásba lépjen ezzel a multiSUMOylated platformmal. Így az RNF4 képes olyan fehérjékhez kötődni, amelyek vagy egyetlen helyen keresztül polSUMOilezettek, vagy több helyen multiSUMOiláltak, és felveti annak lehetőségét, hogy az ilyen multiSIM-multiSUMO kölcsönhatások szélesebb körben elterjedhessenek.
Bevezetés
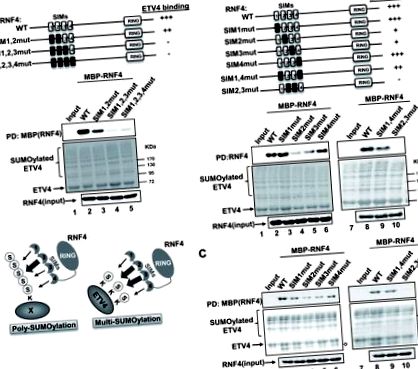
A multimer SUMO kölcsönhatások megértése érdekében megvizsgáltuk az RNF4 SUMOylated ETV4 (más néven PEA3 és E1AF) kötődésének követelményeit. Az ETV4 egy ETS transzkripciós faktor, és egy olyan SUMO módosulási hellyel rendelkező fehérje példája, amely nagy molekulatömegű SUMOilált fajokat képez (Guo & Sharrocks, 2009; Nishida et al., 2007). A SUMOylationről korábban kiderült, hogy fontos az ETV4 transzaktivációs aktivitása szempontjából, de idővel késleltetve kiváltja annak lebomlását is az ERK útvonalon keresztüli növekedési faktor által közvetített szignalizációt követően (Guo & Sharrocks, 2009). Kimutatták, hogy az STUbL RNF4 fontos szerepet játszik mind az ETV4 által közvetített célgén aktiváció, mind az ubiquitin útvonalon keresztüli ETV4 lebontás szabályozásában. Itt megvizsgáltuk, hogy a multiSUMOylation lehet-e eszköz az RNF4 toborzás ETV4-be történő vezetésére. Megkötési vizsgálatok sorozatával megmutatjuk, hogy a multiSUMOylation elősegítheti az RNF4-et tartalmazó multi-SIM fehérje ETV4-hez való kötődését.
Anyagok és metódusok
Plazmid konstrukciók
A következő plazmidokat használtuk emlőssejt transzfekciókban; pSG5-PIAS4 (kódolja a Myc-tagged PIAS4-et) (Frances Fuller-Pace; Jacobs és mtsai., 2007), pCDNA3-Ubc9/UBE2I, pCDNA3-His-SUMO-3 és pCDNA3-His-SUMO-3 (K11R ) (kedvesen szolgáltatta: Ron Hay; Tatham et al., 2001). pAS981 (a pCDNA3 XbaI/KpnI helyeibe klónozott teljes hosszúságú, zászlóval címkézett zebrafish ETV4/PEA3 kódol; Amanda Greenall készítette).
Asztal 1.
| ADS1580 | ATCGGGATCCATGGAGCGGAGGATGAAAGGC |
| ADS1584 | ATCGGAATTCAGTAAGAATATCCACCTCTGTG |
| ADS2581 | GCGGGGAATTCGGTAGTGGTAGTGGTAGTATGTCCGAGGAGAAGCCCAAG |
| ADS2582 | GCGGGCCATGGCTATGTCCGAGGAGAAGCCCAAG |
| ADS2583 | GTGAATCTTTAGAGCCTGCGGCTGCGGACCTGACTCACAATGA |
| ADS2584 | TCATTGTGAGTCAGGTCCGCAGCCGCAGGCTCTAAAGATTCAC |
| ADS2587 | GACTCACAATGACTCTGCTGCGGCTGCTGAAGAAAGGAGAAGGC |
| ADS2588 | GCCTTCTCCTTTCTTCAGCAGCCGCAGCAGAGTCATTGTGAGTC |
Sejtkultúra, ko-immunprecipitációs elemzés és Western-blotolás
A HEK293T sejteket 10% magzati szarvasmarha-szérummal kiegészített DMEM-ben tenyésztettük, és ahol jeleztük, a sejteket 6 órán át forbol-12-mirisztáttal (PMA) (Sigma) (10 nM) kezeltük. A plazmid transzfekciót Polyfect (Qiagen) alkalmazásával hajtottuk végre. Western blot-ot és immunprecipitációt végeztünk az elsődleges antitestekkel; nyúl poliklonális anti-RNF4 (1: 5000 ajándék Jorma Palvino-tól; Häkli és mtsai., 2005), egér monoklonális anti-Flag M2 (1: 2 000 Sigma; F3165) és egér monoklonális anti-MBP (1: 1 000 Abcam, ab49923) < Figure 2A > és 1: 1 000 sejtjelző egér monoklonális macska 2396 < Figure 2B >). A fehérjéket a korábban leírtak szerint detektáltuk (Aguilar-Martinez et al., 2015).
Először megvizsgáltuk, hogy találhatunk-e bizonyítékot az ETV4 multi- vagy poliszumilezésére in vivo. Megvizsgáltuk az ETV4 SUMOylation állapotát a sejtek UBE2I/UBC9 és PIAS4 ko-transzfekcióját követően (a SUMOylation szintek maximalizálása érdekében), vagy vad típusú SUMO3 vagy SUMO3 (K11R) mutánssal. A SUMO ez utóbbi formája nem képezhet poliSUMO láncokat (Tatham et al., 2001). A sejteket PMA-val kezeltük a SUMOylation szintjének fokozása érdekében (Guo et al., 2009). Több nagy molekulatömegű SUMO konjugátumot figyeltünk meg vad típusú SUMO3 jelenlétében (1B. Ábra, 3. és 4. sáv), és azonos konjugációs mintát figyeltünk meg SUMO3 (K11R) jelenlétében (1B. Ábra, 5. és 6. sáv) . A több sáv kialakulása valószínűleg az egyhelyes SUMOylation események és a több hely SUMOylation kombinációjának köszönhető, hogy a nagyobb molekulatömegű alacsonyabb mobilitású fajokat hozzák létre. Tehát, mivel a SUMO3 (K11R) ugyanazt a sávos mintázatot adja, mint a vad típusú SUMO3, a sejtekben megfigyelt több faj valószínűleg a multiSUMOylated ETV4 különböző kombinációit képviseli, nem pedig a polySUMOylated konjugátumokat.
Ezek az ETV4-RNF4 interakciókra vonatkozó megállapítások együttesen azt bizonyítják, hogy a multiSUMOylation platformként működhet egy fehérjét tartalmazó multi-SIM domén toborzásában.
1. ábra nyers adatok. A teljes western blotok láthatók, és az 1. ábra paneljeibe felvett területek kiemelve vannak (négyzetekkel jelölve).
2A. És C. ábra. Nyers adatok. A teljes western blotok láthatók, és a 2. ábra paneljeihez felvett területek kiemelve vannak (négyzetekkel jelölve).
2B. Ábra. Nyers adatok. A teljes western blotokat mutatjuk be, és a 2. ábra paneljeibe felvett területeket kiemeljük (négyzetekkel jelölve).
- Egyenes beszéd a szójáról A táplálékforrás Harvard T.
- Növényi étrend receptek - 9 napos növényi étrend
- Hét ok a tökéletes kiszáradt máj hozzáadásához étrendjéhez - Tápláló világ blog
- Sok imádat Fidóról; s Élelmiszer Seven Hills Állatorvosi Kórház
- A piros fül párosítása csak annyit jelent, hogy könnyedén lefogyj - a BBC