Knomics-Biota - az emberi bél mikrobiota adatainak feltáró elemzésére szolgáló rendszer
Absztrakt
Háttér
Az emberi mikrobiota metagenomikus felmérései egyre szélesebb körben terjednek el az akadémiai kutatásokban, valamint az élelmiszer- és gyógyszeriparban, valamint a klinikai kontextusban. A kísérleti adatok kivizsgálásának intuitív eszközei nagyon érdeklik a kutatókat.
Eredmények
A Knomics-Biota egy webalapú forrás az emberi bél metagenómáinak feltáró elemzéséhez. A felhasználók elkészíthetik és megoszthatják a közös kísérleti sémáknak megfelelő analitikai jelentéseket (például eset-kontroll tanulmány vagy páros összehasonlítás). Az interaktív vizualizációkat és a statisztikai elemzéseket a külső tényezőkkel összefüggésben és a nyilvánosan elérhető, tematikus gyűjteményekbe rendezett több ezer adatállomány összefüggésében nyújtják. A webszolgáltatás a https://biota.knomics.ru címen érhető el.
Következtetések
A Knomics-Biota webszolgáltatás átfogó eszköz az interaktív metagenomikus adatok elemzéséhez.
Háttér
Végrehajtás
A rendszer számítási háttérrendszere a felhőben található (1. kiegészítő fájl: S1. Ábra), és nyilvánosan elérhető szoftveres megoldásokat használ. A webszolgáltatás kezelőfelülete a Yii keretrendszer segítségével valósul meg, az interaktív vizualizációk pedig a d3js könyvtáron alapulnak. A webszolgáltatás a következő címen érhető el: https://biota.knomics.ru. A regisztráció után a felhasználó feltöltheti saját metagenomikus olvasási készleteit (amelyeket 16S rRNS vagy „shotgun”/WGS [teljes genom] szekvenálásával kaptak), valamint adatleíró fájlokat (metaadatokat).
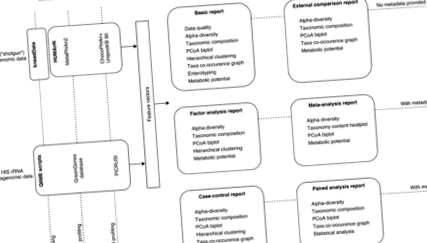
A Knomics-Biota szolgáltatás általános logikája két komponenst tartalmaz: az elsődleges és a másodlagos elemzést (1. ábra). Az elsődleges elemzőkomponens magában foglalja az olvasások alapvető feldolgozását a mikrobiota összetételi profilok megszerzéséhez. A 16S rRNS és WGS mindegyik formátumához az elsődleges elemzési komponens olyan jellemzővektort állít elő, amely magában foglalja a mikrobiális taxonok relatív bőségét a különböző rangokon, valamint a géncsoportokat és az anyagcsere útvonalakat a KEGG Orthology és Enzyme Commission (EC) nomenklatúrái szerint. Ezenkívül egyes funkciókat dedikált módon elemeznek, mivel fontosak az emberi egészség szempontjából - a vitaminok és az SCFA-k szintézise. Ezeket a funkciókat minden mintához kurátus útvonalak segítségével értékelik (2. kiegészítő fájl: S2. Ábra).
A Knomics-Biota webszolgáltatás munkafolyamata. A munkafolyamat két alapvető lépésre oszlik: elsődleges és másodlagos elemzés, mind az amplikon, mind a WGS metagenomikus adatokra
A 16S rRNS adatok elsődleges elemzését QIIME [6] alkalmazásával végezzük, az olvasási szűréstől az OTU-k (operatív taxonómiai egységek) meghatározásáig. A géntartalmat PICRUSt algoritmus segítségével jósolják [7]. A WGS-adatokat a KneadData segítségével elemzik a minőségi szűréshez, és a HUMAnN [8] segítségével - taxonómiai és funkcionális profilozáshoz.
A Python v. 3.2-ben megvalósított másodlagos elemzési komponens magában foglalja a jellemzővektorok statisztikai elemzését (a metaadatokkal együtt, ha vannak ilyenek), statikus ábrák létrehozását, valamint az inputot (JSON formátumban) az interaktív vizualizációs modulokhoz. A másodlagos elemzés munkafolyama a felhasználó által választott jelentés típusától függően változik (lásd 1. ábra).
Az Alapjelentést először minden felhasználói adat számára létrehozzák. Ez magában foglalja a „nyers” adatok minőség-ellenőrzését, a taxonok és funkcionális géncsoportok relatív bőségének, valamint az alfa-diverzitás értékelését. Hierarchikus klaszterezés, enterotipizálás [9] és metabolikus potenciál előrejelzés történik. Az alapvető vizualizációk mellett interaktív modulok állnak rendelkezésre, beleértve a hőtérképet, a PCoA (fő koordináták elemzése) diagramot, az alfa-diverzitás diagramot és az együttes előfordulási hálózatot [10]. A Knomics-Biota Basic és egyéb interaktív jelentései minden modulját a megvalósítás részletei kísérik (algoritmus és használt adatbázisok, a vezérlési paraméterek értéke stb.), Hogy a felhasználó képes legyen függetlenül megismételni az eredményeket - valamint leírni a módszereket a tudományos publikációban.
A szekunder elemzés bioinformatikai algoritmusai között szerepel a PERMANOVA módszer a többváltozós elemzéshez, a regressziós lineáris modellek és az U-teszt a mikrobiális jellemzők és tényezők közötti összefüggések feltárására. A kiugró értékeket Grubbs-teszt segítségével azonosítják és eltávolítják a további statisztikai elemzésekből. A többszörös tesztbeállítás Benjamini – Hochberg eljárással történik.
Eredmények és vita
Számos metagenomikus elemzési csővezetéket fejlesztettek ki. Különböznek az elemzési lehetőségek között - csak az elsődleges „nyers” adatfeldolgozás vagy a haladó lehetőségek biztosításával, lehetővé téve a különböző bemeneti adatformátumokat (16S rRNS szekvenálás vagy WGS adatok). Az összehasonlító adatokat az 1. táblázat tartalmazza, kiemelve, hogy a Knomics-Biota funkciók gazdag repertoárját kínálja, így az alternatíváktól jobb. Mint látható, csak a Knomics-Biota és az MG-RAST [11] szolgáltat publikált metagenómák adatbázisait összehasonlító elemzés céljából. A Nephele [12], valamint a CosmosID és a One Codex platformok hasonló funkciót biztosítanak: „nyers” adatfeldolgozás, fejlett statisztikai elemzés és vizualizációk. Azonban egyikük sem nyújt interaktivitást, amely lehetővé teszi a kijelző menet közbeni paramétereinek megváltoztatását.
A Knomics-Biota tudományos felhasználásra ingyenes. Kereskedelmi felhasználásra speciális engedélyeztetés biztosított. Az ingyenes elemzés ideje függ a várakozási sorban lévő projektek számától, és valószínűleg megváltozik a rendszer fejlődése során, de jelenleg egy tipikus 16S rRNS adatkészlet elemzése, amely körülbelül 100 mintát tartalmaz egyetlen Illumina MiSeq futtatásból (mint elterjedt bemeneti adatformátum) több órán belül feldolgozásra kerül. Összességében a felhasználó körülbelül 5000 16S rRNS-mintát küldhet be egyszerre. Ami a WGS-elemzést illeti, a nagy adatmennyiség és a sor miatt a feldolgozás hosszabb időt vehet igénybe - például 50–100 WGS-metagenómák esetén körülbelül néhány nap.
Mielőtt elkezdené feltölteni saját adatait a Knomics-Biota-ba, lehetőség nyílik a meglévő adatkészletek teljes funkcióinak áttekintésére. Miután névtelenül bejelentkezett egy demo-fiókba, a felhasználó rendelkezésére bocsátják a nyilvánosan hozzáférhető metagenomikus adatokhoz előre kiszámított analitikai jelentéseket, számos nagyszabású vizsgálat metaadatával, amelyek különböző körülmények között vizsgálják a mikrobiómákat, például vastagbélrák [13], gyulladásos bélbetegségek [14 ] és az alultápláltság [15], valamint az étrendi beavatkozásokhoz társítva [3]. A külső adatsorok listáját rendszeresen frissítik az emberi bél mikrobiotájával (valamint egyéb fülkeivel) kapcsolatos újonnan közzétett metagenómákkal.
A regisztráció és a bejelentkezés után a felhasználó létrehozhat egy projektet a fiókjában, és feltöltheti a „nyers” adatokat - a metagenomikus értékeket FASTQ formátumban olvashatja el amplikonon (16S rRNS) vagy WGS-en keresztül. A feltöltés befejezése után a felhasználó folytathatja az elemzést - mindig az Alapjelentéssel kezdve. A többi jelentéstől eltérően az Alap jelentés létrehozása nem igényli sem a metaadatokat, sem a külső kontextus megadását. A jelentés tartalmazza a minőségellenőrzés, a mikrobiota taxonómiai és funkcionális összetétel profilozásának és az alfa-változatosság eredményeit. A meglévő hasonló szolgáltatások gyakran bonyolult konfigurációs lépéseket igényelnek a felhasználótól, csak az alapvető elemzési funkciókat biztosítják [6], vagy erősen specializálódtak [1]. Miután az Alap jelentés sikeresen elkészült, lehetőség van speciális elemzés végrehajtására. A főbb jelentéstípusokat és azok tartalmát röviden az 1. ábra mutatja.
A Knomics-Biota egyik alapvető funkciója az a lehetőség, hogy elemezzük a felhasználói adatokat ugyanazon csővezeték segítségével előre kiszámított, nyilvánosan elérhető cikkek több ezer metagenómájának összefüggésében. A külső adatkészletek gyűjteményét rendszeresen frissítik. A kényelem érdekében témáik szerint gyűjteményekbe (összefüggésekbe) rendezik őket. A fő mikrobiota témák közé tartozik a gyulladásos bélbetegségek (IBD), az étrend, a széklet tömegátültetése (FMT), az antibiotikumok, a világ populációi, a Parkinson-kór és így tovább. Ennek megfelelően, bár a saját adatait összehasonlíthatjuk a Knomics-Biota adatbázis összes metagenómájával, gyakran ésszerű az elemzést a releváns kontextusra korlátozni - a Külső összehasonlító jelentés (felhasználói metaadatok nélkül) vagy a Meta-elemzés jelentés (a felhasználói metaadatok megadva). Az elemzés befejezése után a felhasználót e-mailben értesítjük.
Amikor az egyes minták vagy kontrollcsoportok tagságára vonatkozó információkat feltöltik, elérhetővé válik a megfelelő Case-control jelentés - amely lehetővé teszi ezen adatkészletek statisztikai és vizuális összehasonlítását - hasonlóan a Külső összehasonlítás forgatókönyvéhez. Az interaktív modulok funkcionalitása kibővült, hogy lehetővé tegye a mikrobiota összetételének összehasonlítását a két csoport között. Statisztikai elemzést végeznek a releváns szignifikáns különbségek azonosítására. Az alapvető összetételi jellemzők mellett a bél mikrobiota-specifikus jellemzőit értékelik és összehasonlítják a csoportok között: ezek tartalmazzák a vitaminok és az SCFA-k szintetikus metabolikus potenciálját. A párosított elemzési jelentés az eset-kontroll forgatókönyvhöz hasonló munkafolyamatot tartalmaz, de a párosított adatok (például az ugyanazon alanyoktól kapott antibiotikum-terápia előtt és után nyert metagenómák) figyelembevételével módosították.
Faktoranalízis-jelentés jön létre, ha külső/belső tényezőkkel rendelkező metaadatokat közölnek. A szolgáltatás többtényezős elemzést végez a mikrobiota összetétele és az olyan tényezők közötti jelentős összefüggések azonosítása érdekében, mint az életkor, a testtömeg-index (BMI), a klinikai állapot stb. Ezenkívül egy külön típus - idősor-jelentés - foglalkozik az egymást követő csoportosított minták vizsgálatával, beleértve olyan algoritmusokat is, mint a taxonstabilitás elemzése és ezeknek a pontoknak a vizualizálása.
Az együttműködésen alapuló kutatás megkönnyítése érdekében a Knomics-Biota lehetővé teszi a hozzáférés-ellenőrzés beállítását. Alapértelmezés szerint a feltöltött adatokat és a létrehozott jelentéseket csak a felhasználó láthatja. Lehetséges azonban a jelentések bármelyikének megosztása csak megtekintés módban (állandó link használatával), vagy a projekt privát megosztása a szolgáltatásban regisztrált munkatársakkal.
Következtetések
A Knomics-Biota szolgáltatás kényelmes eszköz a metagenómák együttműködésen alapuló feltáró elemzéséhez a nyilvánosan elérhető adatok összefüggésében. A specifikus betegségek és a világ populációinak mikrobiotájára összpontosító metagenómák tematikus gyűjteményei, az étrendi és orvosi beavatkozások hatása hasznos az összehasonlító felmérésekhez és az adatok validálásához. A bélmikrobiota mellett a rendszer készen áll a metagenómák feldolgozására egy önkényes környezetből, így a bioinformatikai szakértelemmel rendelkező és anélküli felhasználók betekintést nyerhetnek a bonyolult mikrobiális közösségek rendszerbiológiájába.
Elérhetőség és követelmények
Projekt neve: Knomics-Biota.
Operációs rendszer (ek): Platformtól független.
Programozási nyelv: Python.
Egyéb követelmények: böngésző, internetkapcsolat.
Engedély: GNU GPL.
Bármely korlátozás, amelyet nem akadémikusok használhatnak: a tudományos felhasználás ingyenes; kereskedelmi célú felhasználáshoz engedély szükséges.
Rövidítések
Rövid láncú zsírsav
Teljes genom szekvenálás
Hivatkozások
Yarygin KS és mtsai. Resistomap - az emberi bél mikrobiota antibiotikum rezisztomjának online megjelenítése. Bioinformatika. 2017; 33 (14): 2205–6.
Yarygin K, Tyakht A, Larin A, Kostryukova E, Kolchenko S, Bitner V, Aleksejev D. Specifikus géncsoportok bőségprofilozása előre kiszámított bélmetagenómák felhasználásával új biológiai hipotéziseket eredményez. PLoS One. 2017; 12 (4): e0176154.
Klimenko N és mtsai. Mikrobiomaválaszok egy kontrollálatlan rövid távú diétás beavatkozásra a polgári tudományos projekt keretében. Tápanyagok. 2018; 10 (5): 576.
Odintsova V, Tyakht A, Alekszejev D. A mikrobiális összetételre vonatkozó adatok statisztikai elemzésére vonatkozó irányelvek a metagenomikus szekvenálásból következtetnek. Curr kiadások Mol Biol. 2017; 24: 17–36.
Sudarikov K, Tyakht A, Alekszejev D. A metagenomikus adatok vizualizálásának és elemzésének módszerei. Curr. Mol. Számok Biol. 2017; 24: 37–58.
Caporaso JG és mtsai. A QIIME lehetővé teszi a nagy áteresztőképességű közösségi szekvenálási adatok elemzését. Nat Methods. 2010; 7 (5): 335–6.
Langille MGI és mtsai. Mikrobiális közösségek prediktív funkcionális profilozása 16S rRNS marker gén szekvenciák alkalmazásával. Nat Biotechnol. 2013; 8: 1–10.
Abubucker S, Segata N, Goll J és mtsai. Metabolom rekonstrukció a metagenomikus adatokhoz és azok alkalmazása az emberi mikrobiómában. Eisen JA, szerk. PLoS Computat Biol. 2012; 8 (6): e1002358.
Arumugam M és mtsai. Az emberi bél mikrobiomjának enterotípusai. Természet. 2011; 473 (7346): 174–80.
Kurtz ZD és mtsai. A mikrobiális ökológiai hálózatok ritka és összetételileg robusztus következtetése. PLoS Comput Biol. 2015; 11 (5): e1004226.
Wilke A és mtsai. Az MG-RAST metagenomikai adatbázis és portál 2015-ben. Nucleic Acids Res. 2016; 44 (Adatbázis-kiadás): D590–4.
Weber N és mtsai. Nephele: felhőplatform az egyszerűsített, szabványosított és reprodukálható mikrobiómás adatok elemzéséhez. Bioinformatika. 2017; 8 (2017): 1411–3.
Zeller G, Tap J, Voigt AY és mtsai. A széklet mikrobiotájának lehetősége a vastagbélrák korai stádiumú detektálására. Mol Syst Biol. 2014; 10 (11): 766.
Halfvarson J, Brislawn CJ, Lamendella R és munkatársai. Az emberi bél mikrobiomjának dinamikája gyulladásos bélbetegségben. Nature Microbiol. 2017; 2: 17004.
Smith MI, Yatsunenko T, Manary MJ és mtsai. A malawi ikerpárok bélmikrobiómái ellentmondanak a kwashiorkornak. Tudomány (New York, NY). 2013; 339 (6119): 548–54.
NIH HMP munkacsoport. Az NIH emberi mikrobiom projekt. Genome Res. 2009; 19: 2317–23.
Köszönetnyilvánítás
Köszönetet mondunk a Data Laboratory-nak az interaktív modulok fejlesztéséért, a Go4ward a weboldal fejlesztéséért, Dmitry Rodionov és Andrei Osterman (Sanford Burnham Prebys Medical Discovery Institute) az anyagcsere-utak kúrájában nyújtott segítségért.
Finanszírozás
Ezt a munkát az új technológiák kidolgozásával és kereskedelmével foglalkozó központ „Skolkovo” fejlesztési alapja támogatta [# G94/16 a Knomics LLC-hez].
Az adatok és anyagok rendelkezésre állása
Szerzői információk
Hovatartozások
Kutatási és fejlesztési osztály, Knomics LLC, Skolkovo Innovációs Központ, Moszkva, Orosz Föderáció
Daria Efimova, Anna Popenko, Anatolij Vasziljev, Ilja Altukhov, Nyikita Dovidcsenko, Vera Odintsova, Natalja Klimenko, Robert Loshkarev, Maria Pashkova, Anna Elizarova, Viktoriya Voroshilova, Szergej Slavskii, Jurij Pekov, Jekatyerina Filippova, Jevgenyij Alekszova
Számítástechnikai laboratórium, ITMO Egyetem, Szentpétervár, Orosz Föderáció
Alekszandr Tyakht és Dmitrij Alekszejev
Biológiai és Orvosi Fizikai Kar, Moszkvai Fizikai és Technológiai Intézet (Állami Egyetem), Moszkva, Orosz Föderáció
Ilya Altukhov, Maria Pashkova, Anna Elizarova, Viktoriya Voroshilova, Sergei Slavskii, Tatiana Shashkova & Evgenii Levin
Élettudományi osztály, Skolkovo Tudományos és Technológiai Intézet, Moszkva, Oroszország
Biológiai tanszék, Lomonoszov Moszkvai Állami Egyetem, Moszkva, Oroszország
Novoszibirszki Állami Egyetem Citológiai és Genetikai Intézete, Novoszibirszk, Orosz Föderáció
Fehérje Kutató Intézet, Orosz Tudományos Akadémia, Pushchino, Moszkva, 142290, Oroszország
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
A PubMed Google Scholar alkalmazásban is kereshet erre a szerzőre
Hozzájárulások
AT és DA felügyelte a munkát. IA, AV, RL és ND tervezték a webszolgáltatás architektúráját. AV, IA, DE, AT és YP irányította a csapatmunkát. DE, NK, IA, AV, AP, ND, VO, RL, MP, AE, VV, SS, EF, TS és EL fejlesztették ki a szoftvert. NK, DE, ND, MP, AE, VV, SS és EL gyűjtötték, gondozták és feldolgozták az adatokat. AP, AT és DE elkészítette a kéziratot. Minden szerző elolvasta és jóváhagyta a végleges kéziratot.
- Jó zsírok és rossz zsírok adatelemzése - Marty Kendall
- Hogyan alakítja a táplálkozás és az anyai mikrobiota az újszülött immunrendszerét Nature Reviews Immunology
- Lactobacillus Rhamnosus a kemoterápiával kapcsolatos hasmenés megelőzésében - Teljes szöveg nézet
- Hogyan erősítsük meg az emésztőrendszert az ayurvéda - ayurvéda Bansko révén
- Az LCHF használatának megkezdése; 1. rész Tiszta nap Az emberi potenciál tudománya