Mély dokkolás: Mély tanulási platform a struktúrán alapuló kábítószer-felfedezés bővítéséhez
Francesco Gentile
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Vibudh Agrawal
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Michael Hsing
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Anh-Tien Ton
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Fuqiang Ban
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Ulf Norinder
∇ Swetox, Toxikológiai Tudományok Egysége, Karolinska Institutet, Forskargatan 20, SE-151 36 Södertalje, Svédország
Stockholm Stockholm Egyetem Számítástechnikai és Rendszertudományi Tanszék, Box 7003, SE-164 07 Kista, Svédország
Martin E. Gleave
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Artem Cerkaszov
† Vancouveri Prosztata Központ, University of British Columbia, Vancouver, British Columbia V6H3Z6, Kanada
Társított adatok
Absztrakt
Rövid absztrakt
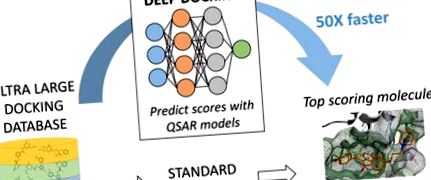
Kidolgoztuk a Deep Docking, egy mély tanulási platformot, amely kvantitatív struktúra-aktivitás kapcsolat modellekre támaszkodik, kiképezve ultratalag nagy adatbázisok kis részeinek dokkoló pontszámával, hogy megjósolják a fennmaradó bejegyzések pontszámát, és ezáltal 50-szeresére gyorsítsák a virtuális szűrést.
Bevezetés
A kábítószer-felfedezés drága és időigényes folyamat, amely számos kihívással néz szembe, többek között az alacsony találati arányú, nagy áteresztőképességű szűréshez. 1,2 A számítógépes gyógyszerfelfedezés (CADD) módszerei jelentősen felgyorsíthatják az ilyen szűrések ütemét, és drasztikusan javíthatják a találati arányokat. 3 A molekuláris dokkolást rutinszerűen használják milliónyi molekulaszerkezetet tartalmazó virtuális könyvtárak feldolgozására számos ismert háromdimenziós szerkezetű gyógyszercélpont ellen.
A rendelkezésre álló vegyi anyagok automatizált szintézisének és fejlődésének legújabb fejleményei nagyszerű lehetőségeket jelentenek a virtuális szűrés (VS) megközelítései általában és különösen a dokkolás terén, de teljesen új kihívásokat is felvetnek. Például a széles körben használt ZINC könyvtár a 2005. évi 700 000 bejegyzésről 4 2019-ben 1,3 milliárd feletti molekulára nőtt 5, ami figyelemre méltó 1000-szeres növekedést jelent. Az ilyen könyvtárak átvilágításában még mindig hiányzik a tapasztalat, és még mindig vita tárgya a kisebb gyűjteményekkel szembeni dokkolás előnye. 6 Úgy tűnik azonban, hogy nemrégiben megjelent művek támogatják a VS kiterjesztését az ultranagy vegyi könyvtárakra. Lyu és munkatársai nemrégiben tett úttörő tanulmányában 7 szerző 170 millió lekéréses molekulaszerkezet dokkolásáról számolt be, amely azt mutatja, hogy az ilyen adatbázisok VS-je lehetővé teszi erősen hatékony inhibitorok, valamint új kémiai osztályok felfedezését, amelyek rutinszerűen nincsenek jelen. átvilágított raktárkönyvtárakat. Később más dokkoló vizsgálatok, amelyekben nagy molekulagyűjtemények vettek részt, hasonló következtetésekhez vezettek. 9,10
Korábban a dokkolási pontszámok előrejelzésének lehetőségét sekély kvantitatív struktúra – aktivitás kapcsolat (QSAR) modelleken keresztül vizsgáltuk (3D „induktív” leírók segítségével 12) és mások, egy támogató vektor gép vagy véletlenszerű erdő felhasználásával konform prediktorokkal. 13,14 Ezen módszerek egyike sem kínál elegendő sebességnövelést a molekulák milliárdjainak kezeléséhez, és az ilyen vizsgálatok így legfeljebb néhány millió vegyületre korlátozódtak. A mély tanulás (DL) viszont különösen alkalmas nagy adatkészletek feldolgozására, 15 és a módszer a hagyományos gépi tanulási technikákhoz képest kiváló teljesítménye miatt gyorsan felkelti az érdeklődést a gyógyszerek felfedezése iránt. 16–18 Így arra számítunk, hogy a DL használata teljes potenciált és valódi szinergiát nyithat meg a dokkoló és a QSAR módszertanok között, és teljes mértékben kihasználja az ultralátagos dokkolási adatbázis adatait.
Eredmények
A jelenlegi tanulmányban bevezettük a gyorsan kiszámított és a céltól független QSAR leírók (például a 2D molekuláris ujjlenyomat), a dokkoló adatbázis iteratív és gyors véletlenszerű mintavételének használatát, és elsősorban a DL alkalmazását megjósolni a még feldolgozatlan adatbázis bejegyzések dokkolási pontszámát minden egyes iterációs lépésnél. Ennek eredményeként a DD akár 100-szoros csökkenést ér el egy ultralarge dokkoló adatbázisban, és akár 6000-szeres gazdagodást is elérhet a legmagasabb rangú találatok esetében, miközben elkerüli a kedvező virtuális találatok jelentős elvesztését, amint azt az alábbiakban tárgyaljuk.
DD csővezeték
Az ultraterjes dokkoló adatbázis (például a ZINC15) minden bejegyzéséhez kiszámítják a ligandum alapú QSAR leírók (például molekuláris ujjlenyomatok) standard készletét;
Megfelelő méretű oktatási részhalmazból véletlenszerűen veszünk mintát az adatbázisból, és a hagyományos dokkolási protokoll (ok) használatával dokkoljuk az érdeklődő célpontba;
A képző vegyületek generált dokkolási pontszámait ezután egy DL modellen keresztül összekapcsoljuk a 2D molekuláris leíróikkal; dokkolási pontszám-levágást (tipikusan negatív) használunk az edzésvegyületek virtuális találatokban (pontozás a cutoff alatt) és nonhits-ben (pontozás a cutoff felett);
A kapott QSAR mély modellt (amelyet empirikus dokkolási pontszámokra oktattak) felhasználják az adatbázis még feldolgozatlan bejegyzéseinek dokkolási eredményeinek előrejelzésére. Ezután egy előre meghatározott számú előre jelzett virtuális találatot véletlenszerűen veszünk mintába, és felhasználjuk az edzéskészlet növelésére;
A b – d lépéseket addig ismételjük, amíg egy előre meghatározott számú iterációt el nem érünk, és/vagy egy ultraterős dokkoló adatbázis feldolgozott bejegyzéseit konvergáljuk.
A DD csővezeték vázlata. (Tetejére) DD inicializálás: egy kis molekulamintát véletlenszerűen kivonnak egy ultratalagú dokkoló adatbázisból, és dokkolnak a vizsgált célponthoz. A létrehozott dokkolási pontszámokat ezután egy QSAR mély modell kiképzésére használják. A létrehozott QSAR megoldást ezután felhasználják az adatbázis fennmaradó részének dokkolási eredményének megjóslására és az előrejelzett virtuális találatok visszaküldésére, amelyek szükségesek az 2. iteráció elindításához. (Alul) DD szűrés: a 2. iterációtól kezdve a mély modell fokozatosan javul a képzés bővítésével állítsa be véletlenszerűen mintavételezett QSAR-előrejelzett virtuális találatokkal az előző DD iterációból (amelyeket a tényleges dokkoláshoz is kiválasztanak). A ciklust megismétlik egy előre meghatározott számú iteráció után, amely után a DD visszajuttatja a legjobb pontszámú molekulákat egy adatbázisból. Ezt a végső könyvtárat utólag lehet feldolgozni a maradék alacsony pontozású entitások eltávolítása érdekében. Alternatív megoldásként a 2–11. Lépéseket végre lehet hajtani egy ultraterjes dokkoló adatbázis konvergenciájáig.
A DD-ben a virtuális találatok visszahívása (vagyis az adatbázisból visszakeresett tényleges virtuális találatok százalékos aránya) implicit módon egy valószínűségi küszöbön keresztül kerül meghatározásra, amely úgy van kiválasztva, hogy a tényleges virtuális találatok 90% -át belefoglalja az érvényesítési készletbe. Ezután ugyanazt a küszöbértéket alkalmazzák a független tesztkészletre, és a virtuális találatok visszahívását értékelik a modell általánosíthatóságának értékelése céljából. Ha az érvényesítés visszahívása és a tesztkészletek összhangban vannak egymással, akkor a modellt az adatbázis összes bejegyzésére alkalmazzák (további részletek a Methods-ban találhatók). Bár a visszahívási értékek kifejezetten jóváhagyhatók voltak például konformális prediktorok alkalmazásával, 14,19 nem figyeltünk meg szignifikáns különbségeket a DD eredő teljesítményében.
A DD-folyamat futtatására szolgáló szkriptek nyilvánosan elérhetők a GitHub-ban, a futtatás beállítására vonatkozó utasításokkal és néhány további eszközzel a HPC-fürtök automatizálásának megkönnyítésére, a https://github.com/vibudh2209/D2 címen.
Ultra nagy dokkoló adatbázis mintavétel
A reprezentatív és kiegyensúlyozott képzési készlet kiválasztása minden modellezési munkafolyamat kritikus lépése. A kémiai tér mintavételének összefüggésében egy megfelelő DD képzési készletnek hatékonyan kell tükröznie az adatbázis kémiai sokféleségét. Számítani lehetett arra, hogy a mintavétel nagyságának növelése és a dokkoló alap előretörése végső soron javítja, vagy akár közelíti a kémiai tér lefedettségét. Másrészt jelenleg kémiai struktúrák milliárdjainak bármilyen módon vagy formában történő csoportosítása nem megvalósítható, és az is bebizonyosodott, hogy a nagy könyvtárak kikötése előtt a kikötés jelentősen csökkentheti az aktív kemotípusok rangját, ezáltal akadályozva az új kemotípusok felfedezését. inhibitorok vagy aktivátorok. 7 Ezenkívül a mintavétel elfogultsága olyan molekulák felé, amelyeket a DD potenciálisan virtuális találatként rangsorol, kizárhatná az alacsony rangú, mégis igaz pozitív molekulákat a modellképzésbe való kiválasztásból; ezért az összes DD iterációhoz véletlenszerű mintavételt választottunk. Végül a DD edzéskészlet méretének (például a tényleges dokkolás mértékének) döntő hatása lenne a számítási futásra, és gondosan ellenőrizni kell.
A képzési készlet mintaméretének hatása a modell általánosíthatóságára. a) A tesztkészlet visszahívások átlagértékei különböző mintanagyságok alapján számítva. Az értékek minden cél esetében megközelítik a 0,90-et, ha az edzéskészlet mérete 250 000 és 1 millió molekula között van. (b) A standard eltérések (STD) megközelítésének 0 variációi 1 millió molekula mintaméretnél. Futtattunk egy iterációt minden célhoz, és ötször megismételtük a számításokat minden mintavételi méretnél.
A ZINC15 méretének csökkentése DD virtuális szűréssel
A DD módszertanának fő célja az, hogy a milliárdnyi bejegyzésből álló ultragyárt dokkoló adatbázist egy olyan kezelhető, néhány millió molekulát tartalmazó részhalmazba redukálja, amely még átfogja a virtuális találatok túlnyomó részét. Ez a végső molekuláris részhalmaz ezután egy vagy több dokkoló program segítségével normál módon dokkolható a célpontba, vagy más VS eszközökkel utófeldolgozható. A DD módszer a mély neurális hálózat (DNN) edzésének iteratív fejlesztésére támaszkodik azáltal, hogy az edzéskészletet minden korábbi iterációból előre jelzett találatmolekulákkal bővíti, miközben a döntő cutoff is fokozatosan szigorodik. Alaposan értékeltük ennek a DD protokollnak a teljesítményét azáltal, hogy a FRIN dokkolóprogram segítségével a ZINC15 összes 1,36 milliárd molekuláját átvilágítottuk a fent bemutatott 12 fehérje célhoz képest. 21 Nevezetesen, maga a DD nem dokkoló motor, hanem egy DL-pontszám előrejelző, amelyet bármilyen dokkolási programmal együtt lehet használni az a priori kedvezőtlen, „nem dokkolható” molekuláris entitások gyors kiküszöbölésére, és ezért drasztikusan megnöveli a tényleges dokkolás sebességét.
A DD hatékonyságának bemutatásához a folyamatot rögzített paraméterkészlettel, például iterációk számával, visszahívási értékekkel és másokkal teszteltük, hogy objektív összehasonlítást nyújtsunk a 12 vizsgált rendszer között. Előre látható, hogy a DD-felhasználók más szimulációs paramétereket akarnak használni, mint a miénk, amelyek leginkább megfelelnek az idő- és erőforrás-allokációjuknak: például kevesebb iteráció több dokkolással iterációnként és kevesebb DL-ciklus lehet optimális választás a sok CPU-val rendelkező fürtök kiszámításához és kevés GPU, és fordítva.
DD teljesítménystatisztika 12 gyógyszercélra. (a) A virtuális találatok kiválasztásához használt pontszám-határértékek variációja minden egyes iterációnál. (b) Az egyes iterációk után virtuális találatként jósolt molekulák számának változása. (c) A dokkolási pontszám átlagértékeinek iteratív javulása véletlenszerűen kiválasztott molekulák esetében, amelyeket az edzéskészlet növelésére használnak. d) 100 legmagasabb rangú jósolt virtuális találatra kiszámított gazdagítási értékek minden egyes iteráció után.
- Mély tanulás - gépi tanulás felhasználása a biológiai látás tanulmányozásához
- A diuretikumok gyógyszerhasználata, típusai, mellékhatásai és kölcsönhatása
- Kontrasztos új súlycsökkentő gyógyszerelőlegek
- Eminem elárulja, hogy a kábítószer-függősége egy csomó testsúly elérésére késztette - élet; Stílus
- Az Eminem elárulja, hogy a drogfogyasztás hogyan hízott meg, gyógyult fel - gördülő kő