Az AMPK aktiválása megvédi a diéta által kiváltott elhízást az Ucp1-független termogenezissel a szubkután fehér zsírszövetben
Alice E. Pollard
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, Egyesült Királyság
Luís Martins
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Phillip J. Muckett
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Sanjay Khadayate
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Aurélie Bornot
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, Egyesült Királyság
Maryam Clausen
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Göteborg, Svédország
Therese Admyre
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Göteborg, Svédország
Mikael Bjursell
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Göteborg, Svédország
Rebeca Fiadeiro
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Laura Wilson
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Chad Whilding
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Vassilios N. Kotiadis
4 Sejt- és fejlődési biológiai tanszék és UCL konzorcium mitokondriális kutatásokhoz, University College London, London, Egyesült Királyság
Michael R. Duchen
4 Sejt- és fejlődési biológiai tanszék és UCL konzorcium mitokondriális kutatásokhoz, University College London, London, Egyesült Királyság
Daniel Sutton
5 Kábítószer-biztonság és anyagcsere, IMED Biotech Unit, AstraZeneca, Babraham, CB22 3AT
Lucy Penfold
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Alessandro Sardini
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Mohammad Bohlooly-Y
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Göteborg, Svédország
David M. Smith
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, Egyesült Királyság
Jon A. Olvassa el
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, Egyesült Királyság
Michael A. Snowden
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, Egyesült Királyság
Angela Woods
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
David Carling
1 MRC Londoni Orvostudományi Intézet, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
6 Klinikai Tudományok Intézete, Imperial College London, Hammersmith Kórház, London W12 0NN, Egyesült Királyság
Társított adatok
Adatok elérhetősége
A tanulmány megállapításait alátámasztó adatkészletek kérésre a megfelelő szerzőtől kaphatók. Az ebben a vizsgálatban felhasznált RNS-szekvencia-adatkészletek a Gene Expression Omnibus (GEO) cégtől kaphatók> GSE120429.
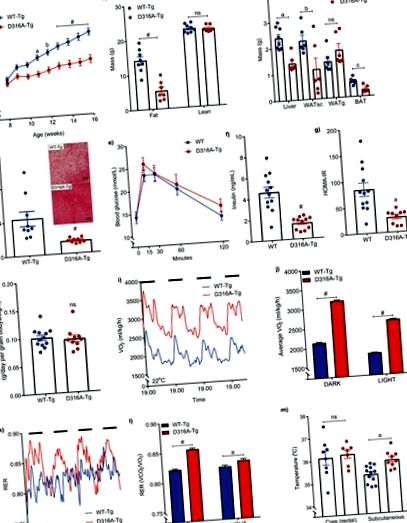
A vad típusú γ1 (WT-Tg) vagy a D316A γ1 transzgént (D316A-Tg) expresszáló hím egereket magas zsírtartalmú étrenddel (HFD) etették 8 hetes kortól. a, Testtömeg (n = 20 WT-Tg esetén és 17 D316A-Tg esetén). a P = 0,002, b P = 0,0006 és # P # P a P = 0,0004, b P = 0,0378, c P = 0,0012, ns = nem szignifikáns). d, A máj triglicerid szintje 16 hétig HFD-vel táplált egerekben (n = 9 genotípusonként; # P # P a P = 0,0012). h, Élelmiszerbevitel 5 napos időszakban (n = 12 WT-Tg esetén és 10 D316A-Tg esetében, ns = nem szignifikáns). én, A teljes test oxigénfogyasztását (VO2) folyamatosan, egy 84 órás időszak alatt figyelték, és (j) átlagos VO2 12 órás világos és 12 órás sötét időszakokban (sötét periódusok, amelyeket a folytonos fekete sávok képviselnek). Az egyes egerek adatpontjait kihagytuk a grafikonból, hogy világosabbá tegyük az átlagértékeket és a hibasávok méretét. # P a P = 0,0086 # P a P = 0,0022, ns = nem szignifikáns). A bemutatott eredmények minden esetben az átlag ± sem. Az a, b, e, j és l panelek statisztikai elemzését kétirányú ANOVA-val végeztük, amelyet Bonferroni többszörös összehasonlító tesztje követett. A c, f, g, h és m panelek statisztikai elemzéseit Student t-tesztjével végeztük, párosítatlan, 2-farkú, Welch-féle korrelációt alkalmazva f-re és g-re. A d panel adatait Mann-Whitney teszttel elemeztük.
RNAseq analízist alkalmaztunk a gén expressziójának változásainak meghatározására a WT-Tg és D316A-Tg egerekből izolált WATsc-ben, 16 hétig HFD-vel táplálva (n = 6 egér/genotípus). a, A vulkán ábrázolása lefelé szabályozott génekkel kék színnel, felfelé piros színnel és változatlan szürke színnel. 1353 gént szabályoztak felül, 1102 gént pedig szabályozatlanul (hajtásváltozás> 1,5). Differenciálisan expresszált géneket (DEG) azonosítottunk (P a P = 0,0026 és b P = 0,0494; ns = nem szignifikáns. f, Vulkán telek mint (a) WATsc-től egér-diétával etetett egerekből (n = 6 egér genotípusonként). 1361 gént szabályoztak felül, 1808 gént pedig szabályozatlanul. Az Atp2a1 és a Ryr1 kiemelve a cselekményen. g, A szignifikánsan dúsított gén ontológiai (GO) annotációkat mutatjuk be a lényegesen felfelé szabályozott génekhez. h, Vázlatos diagram, amely a glikolízisben felfelé szabályozott géneket (pirossal kiemelve) és a TCA-ciklust mutatja a D316A WATsc-ben a chow-étrendet tápláló egerekben. Az e panelen bemutatott eredmények az átlag ± sem és a többszörös t-próbával végzett statisztikai elemzés, több összehasonlításhoz igazítva.
A HFD-n megfigyelt génexpressziós változásokkal ellentétben a chow-étrenddel etetett egerek WATsc transzkriptikus elemzése feltűnően eltérő expressziós mintát tárt fel. Több mint 3000 gén jelentősen megváltozott (hajtásváltozás> 1,5, P 4f ábra). A zsírsav-anyagcserében, a TCA-ciklusban, a mitokondriális ATP-termelésben és a glikolízisben részt vevő gének mind nagyon gazdagodtak a D316A-Tg egerek WATsc-jében (4g. Ábra, h), míg a vázizmokhoz kapcsolódó gének, köztük az Atp2a1 és a Ryr1 -szabályozott (4f. ábra). Összességében ezek az eredmények azt mutatják, hogy HFD-n, de nem chow-étrenden, az AMPK-aktiváció váltást vált ki a WATsc-ben egy vázizom-szerű fenotípus felé. Ezzel szemben chow-étrenden az AMPK aktiválása a katabolikus utakban részt vevő gének expressziójának megváltozásához vezet, beleértve a glikolízist és a TCA-ciklust.
Eredményeink azt sugallják, hogy az AMPK aktiválása növeli a barna-szerű adipociták arányát a WATsc depóban, de ezek a sejtek eltérnek a hagyományos bézs színű adipocitáktól, mivel nem expresszálják az Ucp1-et. Az egerek chow-ról magas zsírtartalmú étrendre történő váltása a transzkripciós válasz markáns változását eredményezi ezekben a barna-szerű sejtekben, ami a vázizmokhoz kapcsolódó gének fokozottabb expressziójához vezet. Javasoljuk, hogy ezek a csontvázizom-szerű AMPK-nak átprogramozott termogén (SMART) adipocitáknak nevezett adipociták egy korábban fel nem ismert sejttípust vagy fenotípust képviselhessenek, amelyet a szokásos szubkután fehér adipociták elérhetnek az AMPK tartós aktiválása során. Elegáns vizsgálatok a származási nyomkövetési technikák alkalmazásával kimutatták, hogy különböző típusú adipociták fejlődhetnek ki ugyanazon progenitorsejtekből, amelyek vázizomsejteket eredményeznek 16, 25, 26. A jelenlegi bizonyítékok arra utalnak, hogy a myocyták és a barna adipocyták egy Myf5 + -prekurzorból származnak, míg a fehér adipocyták egy másik prekurzorból származnak. A legtöbb tanulmány azt mutatja, hogy a bézs színű adipociták egy fehér adipocita prekurzorból (Myf5 -) származnak, amely főként a WATsc depóból származik, és fejlődésük fokozódik a hideg-adaptációra reagálva. .
Mód
Állatok
Metabolikus fenotipizálás
P-guanidinopropionsavval történő kezelés
Bizonyos esetekben az egereket HFD-be helyezték át, és 1 hét múlva hozzáférést kaptak 0,5% β-guanidinopropionsavat és 0,13% szacharint tartalmazó vízhez, vagy csak 0,13% szacharint tartalmazó vízhez. A vizet, a táplálékfelvételt és a testsúlyt a következő 3 hétben ellenőriztük.
Légzés mérése fehér zsírszövet-explantánsokban
Biopsziás lyukasztóval (2 mm) szubkután fehér zsírszövetet (1,5-2 mg) kapunk, XF24 Islet Capture mikrolemezekre helyezzük és vizsgálati táptalajban (szubsztrátmentes DMEM (Sigma D5030), 30 mM NaCl, pH 7,4) inkubáljuk. 25 mM glükóz, 0,5 mM nátrium-piruvát). Az oxigénfogyasztás sebességét Sehaorse XF24 Flux analizátorral mértük oligomicin (5 μM), karbonil-cianid-3-klór-fenil-hidrazon (CCCP, 5 μM) és antimycin (5 μM) egymás utáni hozzáadását követően.
Szövettani elemzés
Western blot elemzés
A szöveteket Ultra-Turrax homogenizátorral 10x tömeg/térfogat jéghideg homogenizációs pufferben 50 mM Tris, 50 mM NaF, 5 mM napirofoszfát, 1 mM EDTA, 0,25 M mannit, 1 mM ditiotreitol, 157 μg/ml benzamidin, 4 μg/ml tripszin inhibitor és 0,1 mM fenil-metil-szulfonil-fluorid. A homogenizátumokat 13 000 x g sebességgel 15 percig centrifugáltuk az oldhatatlan anyagok eltávolítása céljából. Az oldható frakció fehérjetartalmát BCA assay kit (ThermoScientific) alkalmazásával számszerűsítettük. A fehérjéket (összesen 50 μg) SDS-PAGE-val választottuk el, és 100 V feszültségen, 90 percen át polivinilidén-difluorid membránra (Millipore Immobilon-FL) vittük át. A membránokat PonceauS-mal festettük a fehérje-transzfer ellenőrzésére, és 4% (w/v) szarvasmarha-szérumalbuminban (BSA) blokkoltuk 1 órán át szobahőmérsékleten. Eltérő rendelkezés hiányában az elsődleges antitesteket 1: 1000 arányban hígítottuk 4% BSA-t és 0,1% Tween-20-at tartalmazó TBS-ben, és 4 órán át szobahőmérsékleten vagy egy éjszakán át 4 ° C-on inkubáltuk a membránnal. A membránokat alaposan mossuk 0,1% Tween-20-ot tartalmazó TBS-sel, mielőtt megfelelő IRDye szekunder antitesttel (LI-COR Biosciences) inkubálnánk TBS-ben 1 órán át szobahőmérsékleten. A blotokat az Odyssey Imaging System (LI-COR Biosciences) segítségével vizualizáltuk, és az ImageStudio 4.0 segítségével számszerűsítettük.
Antitestek
A következő antitesteket használtuk ebben a vizsgálatban: teljes OXPHOS antitest koktél (Abcam, ab110413); Ckmt2 (Abcam, ab55963); Zászló (Cell Signaling, 14793); Pgc1-a (Abcam, ab54481); Tom-20 (SantaCruz, sc-11415); Serca1 (Abcam, ab109899); Serca2 (Invitrogen, MA3-919); Ucp1 (Abcam, ab 10983); vinkulin (Sigma, V9131).
RNS izolálás
A teljes RNS-t hirtelen fagyasztott szövetből izoláltuk, homogenizálással 1 ml TRIzol-ban (Life Technologies)/100 mg szövet jégen. A mintákat egy éjszakán át -20 ° C-on tároltuk, 10 000 x g-vel 15 percig centrifugáltuk, és a homogenátumot friss csőbe vittük. Kloroformot (400 ml/ml) adunk hozzá, és az elegyet 10 000 x g sebességgel 15 percig szobahőmérsékleten centrifugáljuk. A vizes fázist RNS-mentes Eppendorfba vittük, és abszolút etanolt (0,53x térfogat) adtunk hozzá. Az RNS-t RNeasy Mini centrifugáló oszlopokkal (Qiagen) tisztítottuk. Az RNS-t 50 μl RNáz-mentes H2O-ban eluáltuk és NanoDrop spektrofotométerrel számszerűsítettük. A mintákat szükség esetén -80 ° C-on fagyasztottuk.
RT-qPCR
3 μg RNS-t inkubáltunk 1 μl random hexamerrel (50 ng/μl), 1 μl 10 mM dNTP keverékkel és DEPC H2O-val 10 μl-re. A mintákat 65 ° C-on inkubáltuk 5 percig termikus cikleren. 10 μl cDNS-szintézis keveréket (2 μl 10 x RT puffert, 4 μl 25 mM MgCl2-t, 2 μl 0,1 M DTT-t, 1 μl RNaseOut és 1 μl Superscript II-t (Qiagen) tartalmaz) minden mintához adtunk, és az alábbiak szerint ciklusoltunk: 25 ° C (10 perc), 85 ° C (50 perc) és a csöveket jégre visszük, amíg lehűl. Minden mintához 1 μl RnaseH-t adunk, és 10 percig 37 ° C-on inkubáljuk. A primer linearitás meghatározásához minden mintából 5 μl cDNS-t egyesítettünk és sorozatban hígítottunk, hogy egy standard görbét kapjunk. Minden egyes PCR-reakcióhoz 5 μl cDNS-t adtunk 10 μl 2x SYBR-HiROX (Bioline), 1,6 μl előre (AGCCATCTGCATGGGATCAAA) és fordított (GGGTCGTCCCTTTCCAAAGTG) primer keverékhez és ddH2O-hoz 20 μl teljes reakciótérfogatig. A qPCR lemezt Opticon termikus ciklerrel elemeztük Opticon monitor szoftverrel, hogy minden reakcióhoz c (t) értékeket hozzunk létre.
A génexpresszió számszerűsítéséhez a c (t) érték replikátumokat ellenőriztük a primer hatékonysága (%) és konzisztencia szempontjából. Ezután az egyes mintákhoz kiszámoltuk az átlagos c (t) értékeket, és számszerűsítettük azokat a lineáris egyenletet (y = mx + c) használva, amelyeket korábban a standard görbéből határoztak meg, és amelyek megfelelnek a kérdéses génnek. Az egyenletet x-re oldottuk meg, y-vel mint c (t) értékkel. Ezt az értéket 10x felhasználásával transzformáltuk, mivel a standard görbét logaritmikus skálán generáltuk. Ezt a folyamatot minden gén esetében megismételtük, beleértve a kijelölt háztartási gént is. Hacsak másként nem jelezzük, az összes kísérletet Polr2a-ra normalizáltuk, egy RNS-polimerázra, amelyről kiderült, hogy az étrendi beavatkozások során stabil marad.
A génexpressziót ezután a kísérleti háztartási gén expressziójához viszonyított arányként és a kijelölt kontrollhoz viszonyított változásokként is bemutattuk.
RNS-seq elemzés
Az RNS-t extraháltuk szubkután fehér zsírszövetből (genotípusonként 6 egeret tápláltunk chow-val vagy HFD-vel 16 hétig, egyedileg feldolgoztuk). Mindegyik minta esetében 2 μg RNS 60 μl RNáz-mentes H2O-ban minőségértékelést végeztünk BioAnalyzer RNS kit (Agilent CA, USA) alkalmazásával. A további elemzéshez az RNS Integrity Number (RIN) pontszáma> 7 volt szükséges. Az RNS könyvtárakat TruSeq szálas mRNS Library Prep Kit (Illumina) és standard Illumina protokoll alkalmazásával készítettük el. A könyvtárakat Qubit HS (ThermoFisher) és az Agilent BioAnalyzer segítségével számszerűsítettük a szekvenáláshoz megfelelő koncentrációra. Az indexelt könyvtárakat egyesítettük és szekvenáltuk 1,6 pM végkoncentrációban egy Illumina NextSeq 500 nagy teljesítményű futtatáson, páros végű kémia alkalmazásával, 75 bp olvasási hosszúsággal. A szekvenálási adatokat az Illumina bcl2fastq2-v2.16 alkalmazásával demultiplexeltük. Az olvasások minőségét a FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) segítségével értékelték. Az olvasmányokat feldolgoztuk és leképeztük az mm10 egérgenomra a Bcbio-nextgen keretrendszer 0.9.0 verziójával (https://github.com/chapmanb/bcbio-nextgen). Az alkalmazott aligner STAR 2.4.1d volt, és az illesztés minőségét a QualiMap v.2.1.1 alkalmazással értékeltük.
A differenciálisan expresszált gének azonosítását a DESeq2 alkalmazásával hajtottuk végre. Minőségellenőrzési lépésként elvi komponenselemzést készítettünk a minták csoportosulásának értékelésére a génexpresszió szempontjából. Létrehozták a differenciálisan expresszált gének (DEG) listáját, amelyet Log2-szeres változásként fejezünk ki a kontrollmintához képest (WT-Tg), minden génhez igazítva a P.
A differenciálisan expresszált gének (DEG) elemzése
Statisztikai analízis
Eltérő rendelkezés hiányában a bemutatott adatok az átlag ± sem értéket mutatják. A grafikus ábrázoláshoz és a statisztikai elemzéshez a Graphpad Prism szoftvert (v7) használtuk. Két csoport összehasonlítása céljából az adathalmazokat Student's tesztekkel elemeztük, statisztikai szignifikanciát P értékként definiálva (74K, pdf)
Kiegészítő információk
Köszönetnyilvánítás
Ezt a munkát az UK Medical Research Council finanszírozta (MC_US_A654_0003 támogatás a DC-nek). Az AEP-t a BBSRC-CASE Studenthip Award (BB/L502662/1) finanszírozta. Az LW-t a British Heart Foundation Studentship Award finanszírozta. Ezúton szeretnénk köszönetet mondani a teljes állatfiziológiai csoport (MRC LMS) tagjainak a segítségért.
Lábjegyzetek
Adatok elérhetősége
A tanulmány megállapításait alátámasztó adatkészletek kérésre a megfelelő szerzőtől kaphatók. Az ebben a vizsgálatban felhasznált RNS-szekvencia-adatkészletek a Gene Expression Omnibus (GEO) cégtől kaphatók> GSE120429.
Jelentések összefoglalása
A kutatás tervezésével kapcsolatos további információk a jelen cikkhez kapcsolódó Természetkutatási Jelentések Összefoglalóban találhatók.
Szerző közreműködései
Az AEP, LM, PJM, TA, MB, LW és RF elvégezték az in vivo és in vitro vizsgálatokat. SK, AB és MC elvégezték az RNAseq-t és az elemzést. A CW és a DS segített az immunhisztokémiai elemzésben. Az AS és az MBY segített a metabolikus fenotipizálásban. Az LP és AW segített az egerek fenotípusos elemzésében. Az AEP, LM, WK és MRD elvégezték a sejtek légzésvizsgálatait. Az AEP, LM, AW, DMS, JAR, MAS és DC megtervezte és megtervezte a vizsgálatot. Minden szerző hozzájárult a kézirat elkészítéséhez.
Versenyző érdekek
A szerzők kijelentik, hogy nincsenek versengő érdekeik.
- Alacsony táplálkozású étrend és T-2 toxin expozíció által kiváltott Kashin – Beck betegség állatmodellje -
- Az ősök egészsége, elhízás és törpék - Diet Diet
- Az egészséges táplálkozás előnyei nagyobbak az elhízás magas genetikai kockázatával küzdő embereknél Genetikai
- Alacsony szénhidráttartalmú és magas fehérjetartalmú étrend által kiváltott apoptózis patkánymájban
- Alkoholos és magas zsírtartalmú étrend által kiváltott krónikus hasnyálmirigy-gyulladás egérmodellje