In silico megközelítés a vad típusú p53 funkcióinak helyreállításával járó lehetséges mechanizmus előrejelzésében az R273H mutáns p53-at expresszáló tumorsejtekben kis molekulatömegű vegyületekkel
Ibrahim Malami
1 Gyógyszerésztudományi Kar, Usmanu Danfodiyo Egyetem, Sokoto, Nigéria
Aliyu Mohamed
2 Biokémiai Tanszék, Élettudományi Kar, Ahmadu Bello Egyetem, Zaria, Nigéria
Imaobong C. Etti
3 Farmakológiai és Toxikológiai Tanszék, Uyo Universiti, Uyo, Nigéria
Peter M. Waziri
4 Biokémiai Tanszék, Kaduna Állami Egyetem, Kaduna, Nigéria
Alhassan M. Alhassan
1 Gyógyszerésztudományi Kar, Usmanu Danfodiyo Egyetem, Sokoto, Nigéria
Absztrakt
Bevezetés
Anyagok és metódusok
Számítógépes hardver és szoftver
A fehérje-ligandum dokkoló szimulációt 4,00 GB RAM-os Intel® Core i5 2,5 GHz-en futtatták 64 bites Windows 10 operációs rendszerrel (Acer Inc., New Taipei City, Tajvan). A jelen vizsgálathoz használt bioaktív vegyületek, a kurkumin, a flavokawain B és az alpinetin vegyület 3D felépítését a ChemOffice 5.1 változatának (PerkinElmer, Waltham, MA, USA) felhasználásával rajzoltuk meg, és PDB fájlként mentettük el. A humán R273H mutáns p53 mag domén fehérje oldat-röntgenkristály szerkezete (2bim.pdb, 1,98 Å felbontás) (Joerger és mtsai, 2006 [18]), valamint a DNS-felismerés oldat röntgen kristályszerkezete vad- A p53 típust (2ahi.pdb, 1,85 Å felbontás) (Kitayner et al., 2006 [20]) a Discovery Studio 4.5 (Accelryls, USA) segítségével a fehérje adatbankból (www.pdb.org) találtuk be. A fehérje-ligand dokkoló szimulációt az Autodock szoftvereszközök 4.2-es verziójával (Scripps Research Institute, USA) hajtottuk végre.
Számítási tanulmányok
(i) Fehérje-ligand dokkolás
A fehérje-ligand dokkoló szimulációs vizsgálatot kezdetben Autodock szoftver eszközzel hajtották végre. A vízmolekulákat és más heteroatomokat teljesen eltávolítottuk a kristályfehérjéből, és az összes hiányzó hidrogénatomot hozzáadtuk a kristályfehérjéhez. A ligand pdbqt állományt gasteiger töltetek hozzáadásával és a nem poláros hidrogénatomok eltávolításával készítettük a ligandumokba, miközben töltéseiket egyesítettük szénatomjaikkal. A ligandum gyökereit detektáltuk és meghatároztuk forgatható kötéseiket, miközben a torzióikat a megfelelő számukra állítottuk be. A rácsparaméter-fájlt (gpf) úgy készítettük el, hogy rácstérképeket találtunk 60 × 60 × 60 Å-n xyz rácspontokban, 0,375 Å távolságban, és a rácsdoboz helyzetét az R248A és R273H mutáns p53 fehérjét körülvevő aktív hely maradványokban központosítottuk. xyz-koordinátáival 98,591, 82,612 és 33,169 Å. Molekuláris dokkolási szimulációt alkalmaztunk, 2 500 000 energiaértékeléssel, minden 100 populáció nagyságú ligandum esetében 100 GA-futásra. Lamarckian dokkoló genetikai algoritmust alkalmaztak a molekuláris kötőhelyek azonosítására és a ligandumkötési energia, a gátlási állandó és az intermolekuláris energia előrejelzésére (Morris et al., 2009 [29]). Hasonló protokollt hajtottak végre a vizsgálatban használt minden egyes molekulánál.
(ii) Fehérje-nukleinsavak dokkolása
Eredmények
A fehérje-ligand interakció értékelése
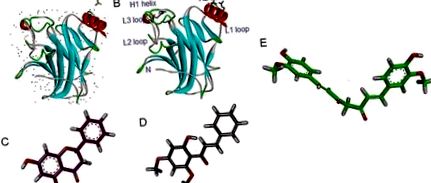
A fehérje-ligandum molekuláris dokkolását szabadenergia és az AMBER erőtér alkalmazásával hajtottuk végre a humán R273H mutáns p53 magdomén kristályszerkezetével szemben (1. ábra (1. ábra)). Ezt követően képesek vagyunk megjósolni a kurkumin, az alpinetin és a flavokawain B kötődését a megcélzott R273H mutáns p53 fehérjéhez. A bioaktív vegyületeket sikeresen dokkolták az R273H mutáns p53 fehérjéhez 36,57, 70,77 és 75,11 µM becsült disszociációs állandóval. Ezenkívül a szabad megkötési energiájukat -6,05, -5,66 és -5,63 kcal/mol értékre becsülték a kurkumin, a flavokawain B és az alpinetin esetében. A végső intermolekuláris energiát és az összes belső energiát -9,04 és -1,62 kcal/mol értékre becsültük a kurkumin esetében. Másrészről az alpinetin és a flavokawain B a becslések szerint -6,52 és -0,43, illetve -7,45 és -0,67 kcal/mol mellett kötődött a p53-hoz.
A bioaktív vegyületek eltérő kötési módját az R237H mutáns p53 fehérje molekuláris kötőfelületén a 3. ábra mutatja (3. ábra). A vegyületeket aminosav oldalláncok veszik körül, a DNS-kontakt csoportokat hordozó p53 fehérje L3 hurok és H2 hélixe között.
A fehérje-DNS kölcsönhatás értékelése
GRAMM programot, valamint a QUASI-DNP, DFIRE és Varani potenciál kombinációját alkalmaztuk a fehérje-DNS molekuláris dokkoló szimuláció értékelésére. A vad típusú p53 DNS-komplex hasított ko-kristályos szerkezetének redocking elemzését kezdetben az NPDock szerveren hajtották végre, hogy teszteljék az alkalmazott fehérje-DNS dokkoló szimulációs vizsgálat pontosságát és hatékonyságát. Következésképpen az Arg248 és Arg273 DNS-kontaktcsoport oldalláncai közvetlen kapcsolatot létesítenek a DNS-foszfát gerinccel. Ezzel egyidejűleg az L3 hurokból a Ser241 oldalláncon keresztül közvetlen kapcsolat is kialakul a DNS-foszfát gerinccel. Ezen túlmenően a H2 spirálból származó Arg280 maradék és az L1 hurok maradékai (Lys120 és Ser121) a guanin bázisokhoz (G8, G3 és G2) és a DNS foszfát gerincéhez kapcsolódnak (4. ábra (4. ábra)).
A redocking elemzésből nyert fehérje-DNS kontaktfelismerés gyakorlatilag megegyezik azzal, amelyet az eredeti ko-kristályosított szerkezetből nyertünk (2ahi.pdb). Ezért a redocking elemzés megerősítette a protein-DNS dokkoló szimulációs vizsgálat érvényességét és hatékonyságát az NPDock szerveren. A fehérje-DNS dokkoló szimuláció során ellenőrzött grafikon-pontszám variációkat az 5. ábra mutatja (5. ábra). .
Ezenkívül lényeges változásokat figyeltek meg a kötőfelület és a DNS-konformáció struktúrájában a bioaktív molekulák bevonásával a vad típusú p53-DNS-komplexhez képest a 7. ábrán (7. ábra). Az R237H mutáns p53-DNS komplex molekulakötő zsebében megfigyelt egyes bioaktív vegyületek kötési módja szerkezetileg azonos. A mutáns p53 kötődési módja azonban a DNS-kötő felülethez lényegesen különbözik az összes komplex között. Ezenkívül a hidrofóbicitásban enyhe változások figyelhetők meg a p53 R273H mutáns szerkezetében is, az összes vegyületet érintve.
- 7 lehetséges ok, amiért feldúltad az álmokat a nőknek; s Egészség
- Agytudományok ingyenes teljes szövegű mélyagyi stimuláció - lehetséges kezelési stratégia patológiailag
- A macska fogyásának 9 lehetséges oka - macska
- Az elhízás funkcionális gyógyászati megközelítése 10 módszer a fogyáshoz; Tartsa ki - UltraWellness
- Új megközelítés a gasztroparézis kezelésében